Una nueva forma de clasificar las pequeñas mutaciones en el ADN ha permitido detectar tumores con errores en la reparación de su material genético. Estos tumores, que responden mejor a la inmunoterapia, ahora pueden identificarse con mayor precisión gracias a un algoritmo llamado PRRDetect.
El estudio, publicado en abril de 2025 en la revista Nature Genetics, estuvo liderado por investigadores del Early Cancer Institute de la Universidad de Cambridge. El trabajo contó con la colaboración de científicos del National Institute for Health and Care Research (NIHR) y de Cancer Research UK, así como con el uso de datos genómicos del 100,000 Genomes Project impulsado por Genomics England.
Esta alianza permitió analizar miles de tumores con tecnologías avanzadas de secuenciación para desarrollar nuevas herramientas de clasificación genética con potencial clínico.
“La secuenciación genómica es ahora mucho más rápida y barata que nunca. Nos estamos acercando al punto en el que secuenciar su tumor será tan rutinario como un escáner o un análisis de sangre”, dijo la profesora Serena Nik-Zainal del Early Cancer Institute de la Universidad de Cambridge, autora principal del estudio.
Una redefinición de las mutaciones abre nuevas posibilidades en oncología
Las inserciones y deleciones (InDels) representan la segunda forma más común de variación genética, pero hasta ahora habían sido difíciles de clasificar con precisión. Los sistemas previos, como COSMIC-83, agrupaban muchas de estas mutaciones en categorías demasiado generales, lo que dificultaba distinguir su origen biológico.
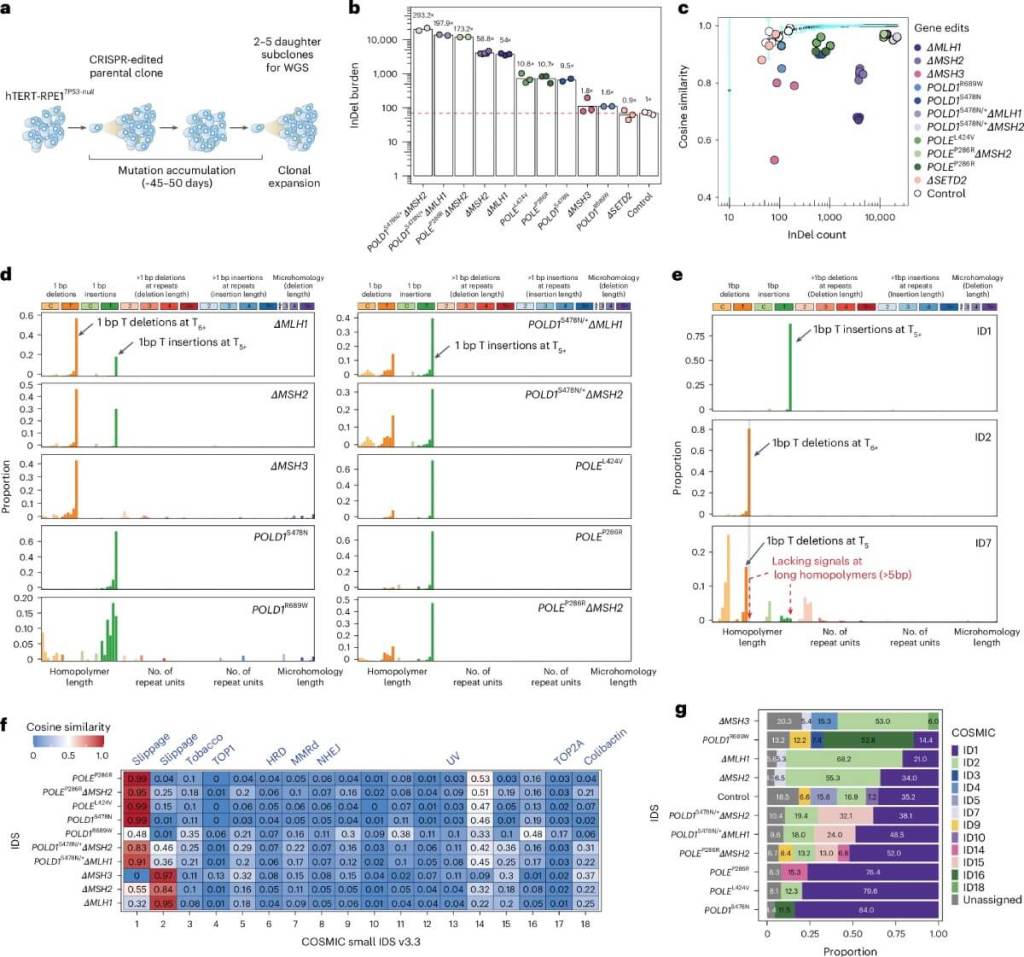
El nuevo estudio propone una clasificación más detallada, basada en el contexto de secuencias flanqueantes y patrones repetitivos, que permite identificar 89 subtipos distintos de InDels.
Esta nueva taxonomía permite identificar firmas mutacionales específicas asociadas a fallos en la reparación post-replicativa del ADN (PRRd), un defecto frecuente en varios tipos de cáncer. Para desarrollar esta clasificación, los investigadores generaron modelos celulares humanos editados por CRISPR y analizaron sus perfiles mutacionales tras 45 días de cultivo. Compararon las mutaciones de las células con ediciones genéticas con las de células no editadas para identificar patrones específicos.
Los resultados mostraron diferencias claras entre mutaciones generadas por errores de replicación normales y aquellas provocadas por disfunciones en los sistemas de reparación del ADN.
Estas diferencias eran casi imposibles de detectar con la clasificación antigua. Al aplicar la nueva taxonomía, los científicos pudieron separar firmas asociadas a mutaciones en polimerasas de ADN de aquellas asociadas a deficiencias en genes como MLH1 o MSH2.
PRRDetect: un algoritmo para encontrar tumores vulnerables
“Para usar la genómica de manera más efectiva en la clínica, necesitamos herramientas que nos den información significativa sobre cómo el tumor de una persona podría responder al tratamiento. Esto es especialmente importante en los cánceres donde la supervivencia es peor, como el cáncer de pulmón y los tumores cerebrales”, explicó Nik-Zainal.
Usando esta nueva clasificación, los investigadores desarrollaron PRRDetect, un algoritmo capaz de identificar tumores con errores en la reparación del ADN.
El sistema se entrenó con datos genómicos de 4.775 tumores pertenecientes a siete tipos de cáncer: colon, endometrio, estómago, vejiga, cerebro, pulmón y piel. PRRDetect utiliza tanto firmas de InDels como de sustituciones de bases (SBS) para clasificar a los tumores según su estado PRRd.
Los tumores con PRRd son más susceptibles a tratamientos con inmunoterapia, ya que sus errores genéticos los hacen más visibles para el sistema inmunitario. En comparación con otros métodos como MSIseq o MMRDetect, PRRDetect demostró tener mayor sensibilidad y especificidad para identificar estos tumores.
Incluso fue capaz de detectar casos sin mutaciones conductoras conocidas, que los métodos tradicionales no habrían identificado.
PRRDetect mostró una precisión superior al evaluar cohortes adicionales de más de 1.300 tumores independientes, con un área bajo la curva (AUROC) de 1.0 y un valor predictivo muy alto. Esta herramienta podría transformar la medicina personalizada, guiando a los oncólogos hacia decisiones terapéuticas más informadas y adaptadas al perfil genético de cada paciente.
Nuevas firmas mutacionales en tumores humanos
La aplicación de la nueva taxonomía a tumores reales permitió descubrir 37 firmas distintas de InDels, muchas de las cuales eran desconocidas hasta ahora. De estas, 10 estaban asociadas a causas ya conocidas, como el tabaco o la exposición a rayos ultravioleta. Otras ocho estaban directamente vinculadas a disfunciones en la reparación post-replicativa, y 19 eran completamente nuevas.
Cada tipo de cáncer mostró una combinación única de firmas mutacionales, lo que sugiere que cada tumor tiene una huella genética propia que puede aprovecharse para su diagnóstico o tratamiento.
Por ejemplo, algunas firmas se observaron exclusivamente en tumores colorrectales expuestos a colibactina, mientras que otras aparecieron en muestras tratadas previamente con quimioterapia basada en platino.
Estas firmas ofrecen pistas sobre mecanismos de daño celular poco conocidos, incluyendo posibles efectos de enzimas como APOBEC o errores inducidos por fallos en las polimerasas de ADN. Algunas firmas nuevas también se encontraron en tumores con alta carga mutacional, pero sin causa evidente, lo que abre nuevas líneas de investigación en biología del cáncer.
“Los cánceres con reparación de ADN defectuosa tienen más probabilidades de ser tratados con éxito. PRRDetect nos ayuda a identificar mejor esos cánceres y, a medida que secuenciamos más y más cánceres de forma rutinaria en la clínica, en última instancia podría ayudar a los médicos a adaptar mejor los tratamientos a los pacientes individuales”, dijo Nik-Zainal.
Implicaciones clínicas y futuro de la medicina genómica
La detección de tumores con PRRd puede cambiar la forma en que se seleccionan pacientes para inmunoterapia, mejorando la eficacia de los tratamientos. Mientras que herramientas como el TMB (tumor mutational burden) se utilizan actualmente para guiar decisiones terapéuticas, PRRDetect ha demostrado que muchas muestras con alta carga mutacional no presentan verdaderas disfunciones PRRd. Esto podría evitar tratamientos innecesarios o ineficaces.
La nueva herramienta permite identificar tumores vulnerables incluso en tejidos poco estudiados, como el sistema nervioso central, vejiga o estómago, donde las deficiencias PRRd también están presentes, aunque con menor frecuencia.
En estos casos, PRRDetect podría revelar oportunidades terapéuticas que antes pasaban desapercibidas. También detecta casos sin mutaciones identificables, lo que sugiere que las firmas mutacionales pueden ser más fiables que los drivers conocidos.
Este estudio refuerza el valor de la genómica como herramienta clínica, más allá de su uso investigativo. Con secuenciación más rápida y asequible, podría convertirse en una prueba rutinaria para personalizar tratamientos y mejorar la supervivencia de pacientes con cáncer.

Una herramienta que redefine el futuro del tratamiento del cáncer
La forma en que se clasifican las mutaciones influye directamente en la capacidad de entender el cáncer, y este estudio demuestra cómo una mejora en esa clasificación puede tener un impacto clínico real. Al identificar con precisión firmas asociadas a defectos en la reparación del ADN, PRRDetect se posiciona como una herramienta poderosa para guiar la inmunoterapia.
Este avance representa un paso clave hacia una medicina oncológica de alta precisión, capaz de adaptar el tratamiento a las vulnerabilidades moleculares específicas de cada tumor. La detección de 27 nuevas firmas mutacionales sugiere que hay aún mucho por descubrir en el genoma del cáncer.
Con PRRDetect, la genómica deja de ser una promesa futura y se convierte en una herramienta concreta para mejorar el diagnóstico, orientar terapias y salvar vidas.
Referencias
- Koh GCC, Nanda AS, Rinaldi G, et al. A redefined InDel taxonomy provides insights into mutational signatures. Nat Genet. (2025). doi:10.1038/s41588-025-02152-y.
Cortesía de Muy Interesante
Dejanos un comentario: