Durante décadas, los científicos han intentado encontrar una manera segura de modificar la actividad de genes concretos sin interferir en el resto del cuerpo. En la práctica, esto ha sido como intentar tocar una sola cuerda en un piano que vibra entero. Cualquier intento de encender o apagar un gen en un tipo específico de célula con frecuencia acababa activando otros genes en otras partes, como un dominó biológico con efectos secundarios no deseados.
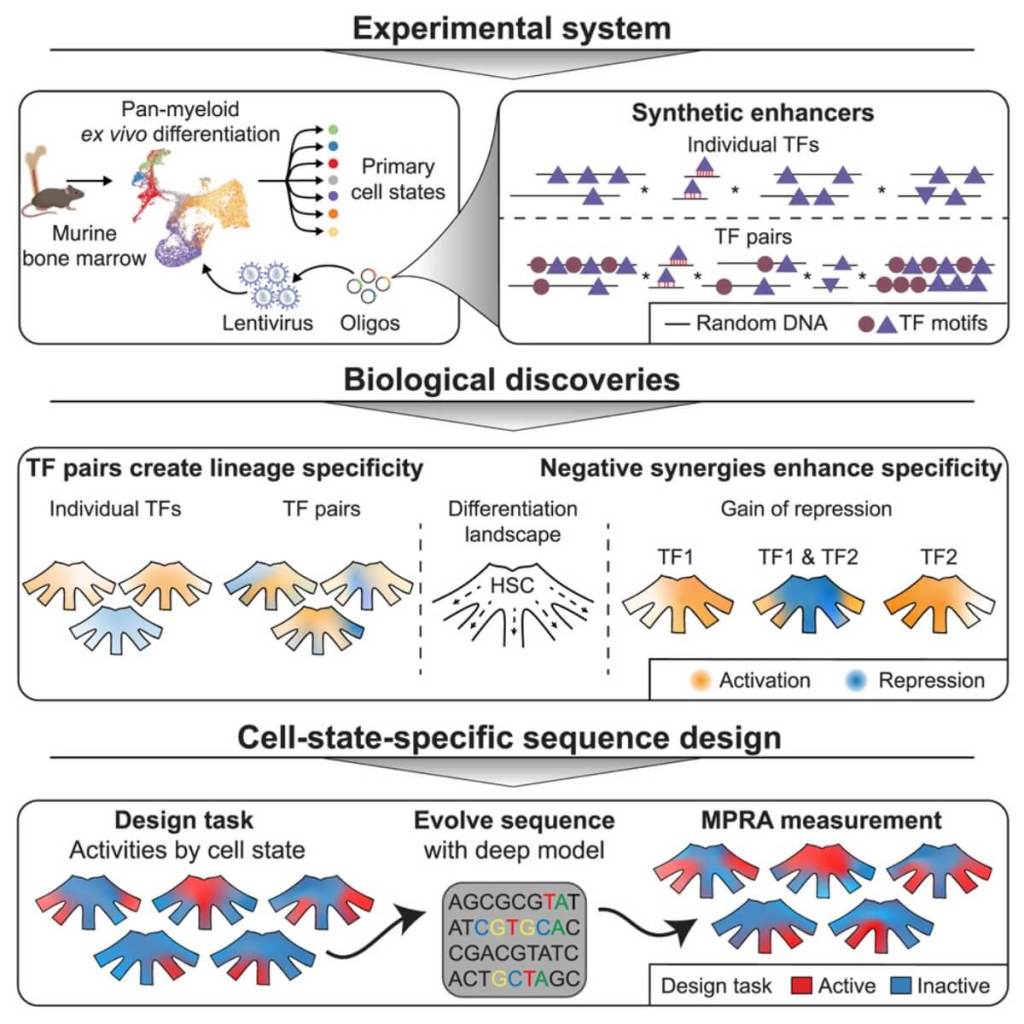
Pero ahora, un equipo de investigadores en Barcelona ha dado un paso crucial: han diseñado fragmentos de ADN artificial capaces de controlar la actividad de genes con una precisión inédita, usando para ello inteligencia artificial y células sanas. El estudio, publicado en Cell y firmado por el grupo de Lars Velten en el Centre for Genomic Regulation (CRG), no solo demuestra que esta tecnología es viable, sino que abre la puerta a tratamientos personalizados más seguros y eficaces.
Un nuevo lenguaje para hablar con las células
La clave de este avance está en lo que los científicos llaman “enhancers”, elementos reguladores del ADN que actúan como interruptores. Estos fragmentos determinan cuándo y dónde se activa un gen, algo esencial para que las células realicen sus funciones específicas. El reto es que, hasta ahora, la mayoría de estudios sobre enhancers se hacían con células cancerosas, porque son más fáciles de manipular. En este caso, los investigadores trabajaron con células madre sanguíneas sanas de ratón, un modelo mucho más representativo de la biología real.
Durante cinco años, el equipo sintetizó más de 64.000 fragmentos de ADN diseñados desde cero, insertándolos en células para observar cómo cambiaban la expresión de los genes. Lo hicieron usando una técnica llamada lentiMPRA, que permite medir la actividad de múltiples fragmentos al mismo tiempo. Esto les dio una base de datos enorme con la que entrenar un modelo de inteligencia artificial capaz de predecir qué secuencias de ADN provocan una activación o una represión genética concreta.
Activadores que también pueden apagar genes
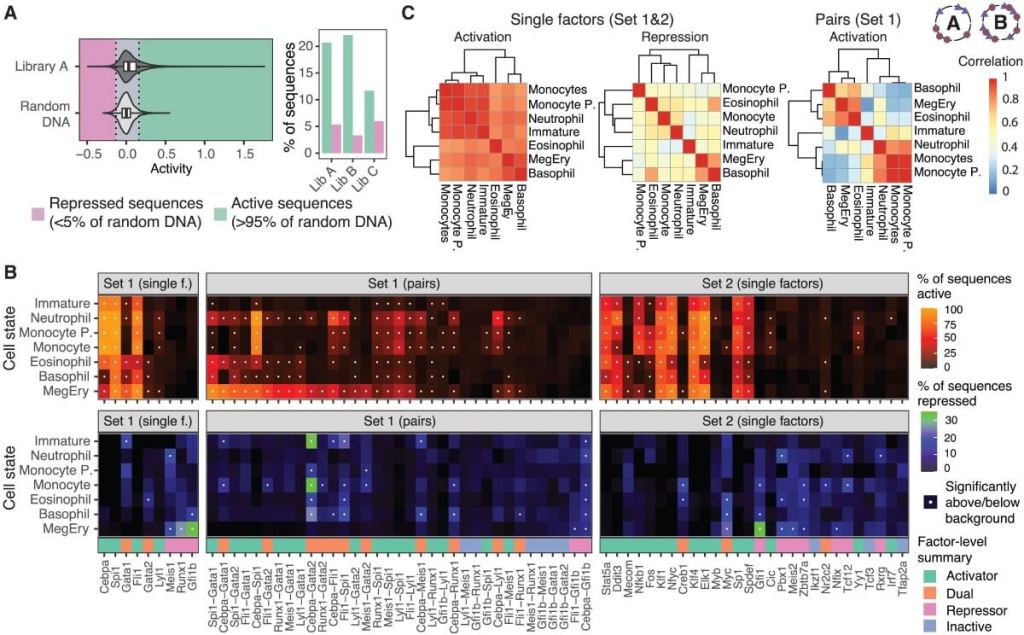
Uno de los hallazgos más sorprendentes fue que los mismos factores de transcripción pueden actuar como activadores o como represores, dependiendo del contexto celular o de cómo se combinen con otros. Según los autores, “las combinaciones de sitios de unión para factores activadores daban lugar con frecuencia a funciones represoras en ciertos estados celulares”.
Esto rompe con la idea tradicional de que un mismo factor siempre tiene el mismo efecto. Los investigadores descubrieron que las interacciones entre factores pueden anularse o incluso invertir su función, en lo que ellos denominan “sinergias negativas”. Por ejemplo, dos factores que por separado activan un gen, pueden juntos hacer que ese mismo gen se apague. Esto permite diseñar fragmentos de ADN que se comporten como interruptores altamente selectivos, solo activos en células específicas.

Una inteligencia artificial entrenada con biología real
Para lograr esta precisión, el modelo de IA fue alimentado con más de 445.000 mediciones de actividad genética en distintos tipos celulares. No se trataba solo de saber si un fragmento funcionaba o no, sino de entender cómo y por qué. El equipo analizó la influencia del número de sitios de unión, su afinidad, orientación, espaciado y combinación. En palabras del artículo: “los motivos de dos factores diferentes pueden interactuar de forma no aditiva”, es decir, la combinación de motivos no sigue una lógica simple de suma de efectos, sino que emergen comportamientos nuevos.
Esto permitió entrenar un sistema capaz de diseñar, desde cero, enhancers que se activan únicamente en ciertas combinaciones de estados celulares. Los investigadores definieron hasta 64 patrones distintos de actividad deseada y lograron generar secuencias que los cumplían. Luego sintetizaron y probaron esas secuencias, con un alto grado de éxito. En más del 75 % de los casos, las secuencias diseñadas por la IA se comportaron tal y como el modelo había predicho.
Aplicaciones en terapias génicas y medicina personalizada
Este enfoque tiene un enorme potencial para la medicina. Uno de los mayores obstáculos de las terapias génicas actuales es la dificultad para dirigir la intervención solo a las células afectadas, sin modificar otras partes del cuerpo. Esta tecnología permitiría diseñar fragmentos genéticos que actúan solo en el tipo celular deseado, como un tratamiento de precisión quirúrgica.
Además, abre la puerta a intervenir en enfermedades causadas por errores en la regulación genética, incluso cuando no hay una proteína concreta a la que dirigir un fármaco. Según el estudio, esto puede ser especialmente útil en trastornos del desarrollo, cánceres o enfermedades autoinmunes, donde la regulación de los genes es tan importante como los genes en sí mismos.
El papel de la ciencia española en la biología sintética
Es destacable que este avance tenga sello español. El trabajo fue liderado desde el CRG en Barcelona, con participación de varios centros vinculados a la Universitat Pompeu Fabra y la colaboración del Barcelona Collaboratorium. En palabras del estudio, “aprovechamos este principio para crear automáticamente potenciadores con especificidad para combinaciones de estados celulares definidos por el usuario”, lo que demuestra la capacidad de la ciencia local para liderar innovaciones globales en campos punteros como la biología sintética y la inteligencia artificial.
El estudio también enfatiza la importancia de usar células primarias, no solo líneas tumorales. Muchos de los principios que descubrieron —como la pérdida de función represora en células de leucemia frente a progenitores sanos— no habrían sido detectables en los modelos tradicionales, lo que refuerza la relevancia de su enfoque experimental.
Mucho más que prueba de concepto
Aunque este estudio es aún un punto de partida, sus implicaciones son profundas. No solo se ha probado que es posible diseñar enhancers con precisión de cirujano, sino que se ha demostrado cómo debe entrenarse una IA para hacerlo bien. Como explican los autores, “este principio permite que los potenciadores detecten proporciones de factores de transcripción y así activen la expresión genética de forma específica para cada estado celular”.
En definitiva, ya no se trata solo de editar genes, sino de programar instrucciones completas sobre cuándo, dónde y cómo deben actuar. Es un paso más hacia una biología computacional donde las células no solo se modifican, sino que se escriben con un lenguaje propio y bien comprendido.
Referencias
- Frömel, R., Rühle, J., Bernal Martínez, A., Szu-Tu, C., Pacheco Pastor, F., Martínez-Corral, R., & Velten, L. (2025). Design principles of cell-state-specific enhancers in hematopoiesis. Cell, 188(1), 1–17. https://doi.org/10.1016/j.cell.2025.04.017.
Cortesía de Muy Interesante
Dejanos un comentario: