Hay bacterias que cazan a otras bacterias. Suena a ciencia ficción, pero es una estrategia real en el mundo microscópico. Una de las más estudiadas es Bdellovibrio bacteriovorus, un microorganismo que se comporta como un auténtico depredador: se lanza sobre sus presas, penetra su membrana y se alimenta de su contenido. Ahora, un nuevo estudio ha revelado que esta bacteria posee una proteína muy peculiar que podría cambiar cómo entendemos sus capacidades.
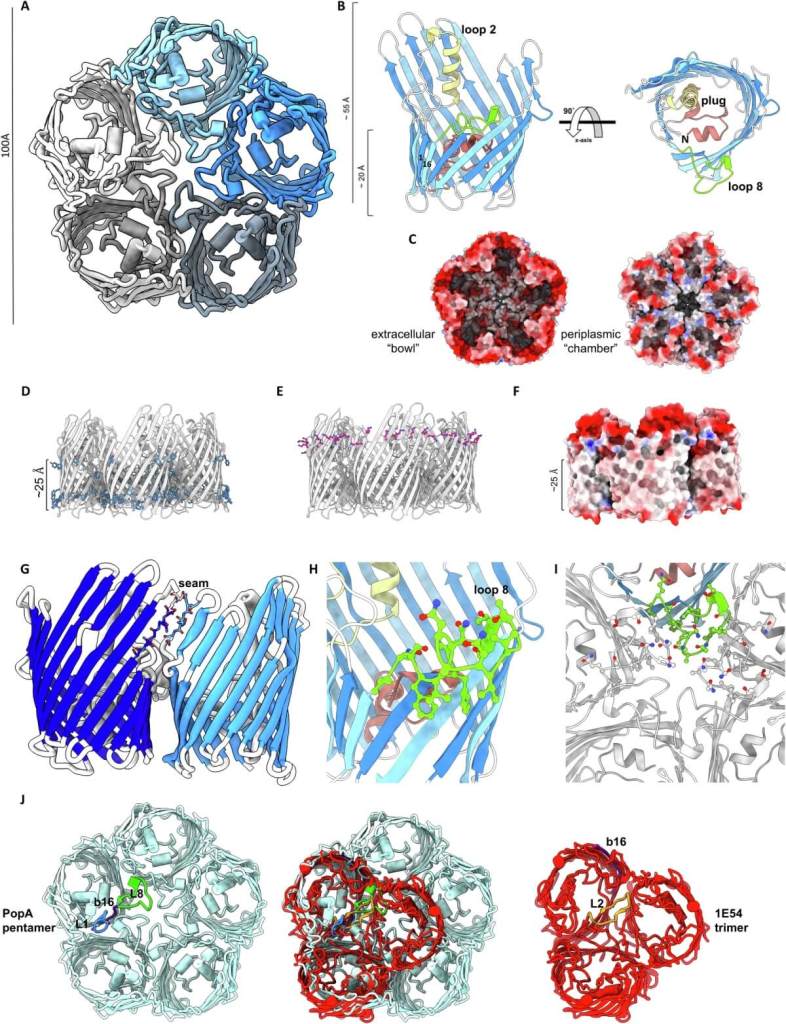
La investigación, publicada en Nature Communications, describe una proteína llamada PopA. Lo más llamativo es que forma una estructura completamente distinta a las conocidas hasta ahora. En lugar de los típicos cilindros simples o tríos, PopA se organiza en un anillo de cinco unidades que actúa como una trampa molecular de lípidos. Este hallazgo no solo cambia lo que sabíamos sobre la arquitectura de estas proteínas, sino que también abre nuevas vías para entender y posiblemente manipular las membranas bacterianas.
Una proteína que no encaja en el modelo clásico
Las proteínas de membrana externa, conocidas como OMPs, tienen funciones esenciales en las bacterias: sirven como canales, sensores o puntos de adhesión. Suelen tener una estructura llamada barril beta, muy conservada y repetitiva. PopA rompe completamente ese patrón. Según los autores del estudio, “la principal proteína de membrana externa de Bdellovibrio bacteriovorus, PopA, difiere de esta arquitectura, formando una superestructura pentamérica parecida a una porina”.
Esta proteína no forma un tubo cilíndrico simple, sino un cuenco con una cavidad central que atrapa lípidos, es decir, componentes grasos que forman parte de la membrana. Esta característica no es decorativa: podría estar relacionada con la capacidad de la bacteria de introducirse en su presa, dañando su membrana o incluso ayudando a absorber nutrientes. La estructura fue visualizada con gran detalle gracias al uso combinado de cristalografía de rayos X y microscopía crioelectrónica, dos técnicas avanzadas para observar biomoléculas.
Además, la cavidad central está tapada por un dominio alfa-helicoidal que actúa como un “tapón”, impidiendo el paso de otras moléculas. Este tipo de organización no se había observado antes en proteínas bacterianas de este tipo, lo que subraya su singularidad.
Daños en la membrana: ¿arma o herramienta?
Uno de los experimentos clave consistió en introducir PopA en bacterias E. coli. El resultado fue contundente: la proteína provocó daños visibles en la membrana celular, algo que no ocurrió con proteínas más comunes. De hecho, los autores observaron que un porcentaje significativo de células comenzaban a formar bultos o deformaciones, especialmente en la membrana interna. Este comportamiento sugiere que PopA podría tener un papel activo en el proceso de ataque de Bdellovibrio a sus presas.
En palabras del artículo: “al introducirse en las membranas de Escherichia coli, [PopA] causa defectos”, lo cual refuerza la hipótesis de que participa en la agresión bacteriana. Este efecto ha sido documentado con microscopía de fluorescencia en células vivas, lo que aporta una prueba visual directa del fenómeno.
Aunque los investigadores no lograron eliminar por completo el gen que codifica PopA sin afectar la viabilidad de la bacteria, el intento fallido refuerza la idea de que esta proteína es esencial, al menos en ciertas condiciones. Su posible papel como “arma molecular” dentro del ciclo de ataque predador merece más investigación.
Una superfamilia bacteriana escondida a simple vista
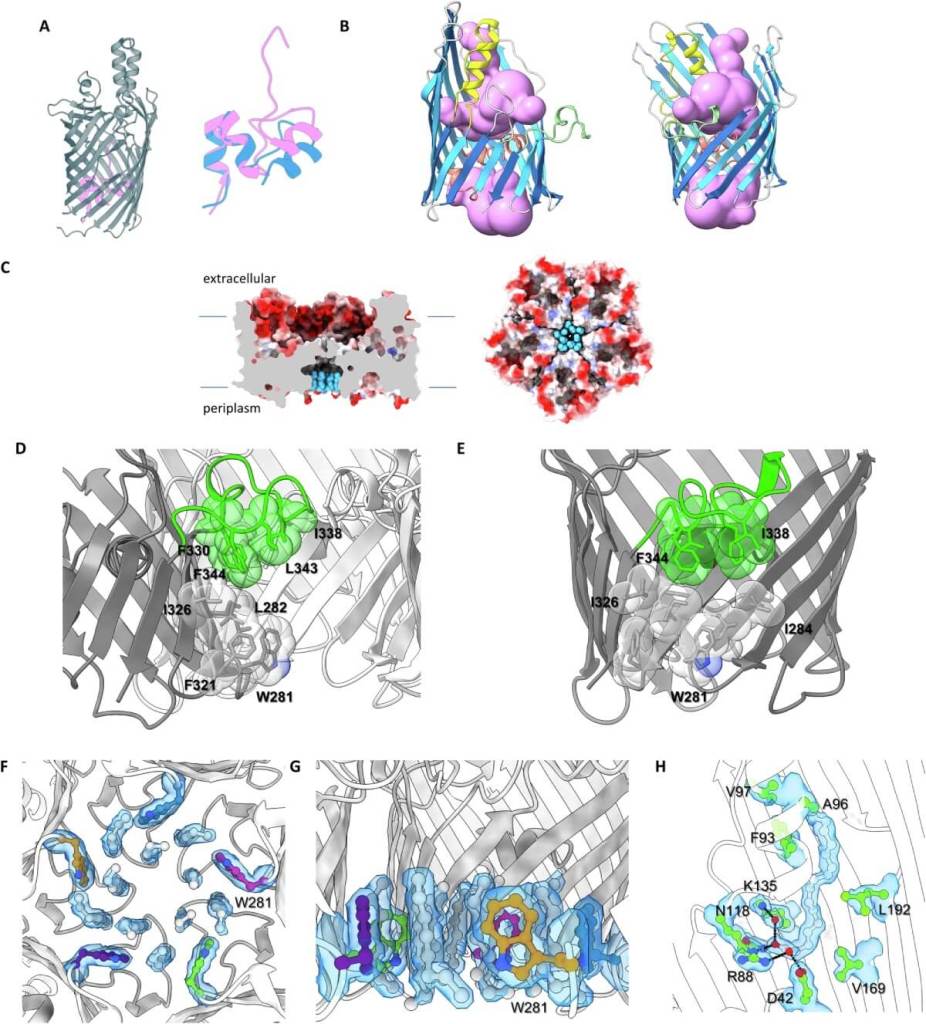
Uno de los hallazgos más impactantes del estudio es que PopA no está sola. Gracias a técnicas de búsqueda estructural asistidas por inteligencia artificial, los investigadores descubrieron que existen proteínas similares en muchas otras bacterias. Algunas adoptan formas de anillos de cuatro (tetrameros), seis (hexameros) e incluso nueve unidades (nonameros), todas compartiendo la capacidad de atrapar lípidos.
Estas estructuras, al igual que PopA, están formadas por barriles beta que se ensamblan en anillos y que generan cámaras internas cargadas de propiedades químicas muy específicas. Lo sorprendente es que muchas de estas proteínas aparecen en bacterias que no son predadoras, lo que sugiere funciones diversas y una historia evolutiva mucho más rica de lo que se pensaba.
El estudio documenta también una segunda familia de proteínas completamente diferente en secuencia, pero con una arquitectura similar, lo que sugiere que la formación de cámaras internas podría ser una estrategia común entre bacterias para manipular lípidos. La hipótesis de los autores es clara: estas proteínas podrían permitir a las bacterias reorganizar sus membranas o interactuar de forma más sofisticada con su entorno.
Trampas moleculares con aplicaciones futuras
El hecho de que estas proteínas puedan capturar lípidos no es un detalle menor. En muchos organismos, los lípidos están involucrados en señalización celular, transporte de moléculas o mecanismos de defensa. La capacidad de atraparlos selectivamente podría ser utilizada por la bacteria para robar componentes a otras células, o incluso para protegerse.
Más allá del interés biológico, este hallazgo tiene implicaciones importantes para la biotecnología. Las proteínas como PopA podrían inspirar el diseño de nanomateriales capaces de capturar o liberar sustancias de manera controlada. Además, comprender su funcionamiento podría dar lugar a nuevas estrategias antibacterianas, basadas en bloquear estas estructuras o aprovecharlas para transportar compuestos activos.
Los autores sugieren que “la estructura de PopA permite estabilidad en ambas membranas, interna y externa, y la cámara lipídica central parece ser un rasgo conservado”. Este tipo de diseño, tan inusual como eficiente, ofrece una ventana fascinante a mecanismos que hasta ahora estaban fuera del radar de la ciencia.
¿Un nuevo modelo para la evolución bacteriana?
El descubrimiento de PopA y sus parientes abre una pregunta más profunda: ¿cuántas otras proteínas pasan desapercibidas simplemente porque no se ajustan a los modelos clásicos? La capacidad de las bacterias para evolucionar soluciones estructurales nuevas parece más amplia de lo que se pensaba, y eso puede obligar a revisar libros de texto.
Al observar cómo se organizan estas proteínas, se ha visto que ciertos elementos estructurales, como el loop 8, son claves para formar las cavidades internas. Esta región actúa como punto de ensamblaje y podría ser determinante en la función de la proteína. Las simulaciones por dinámica molecular confirmaron que las cavidades pueden retener moléculas lipídicas durante largos periodos, lo que les da una funcionalidad estable y específica.
Además, los experimentos demostraron que estas proteínas pueden insertarse en diferentes tipos de membranas, algo poco habitual en este tipo de estructuras. Esta flexibilidad sugiere que podrían haber surgido para adaptarse a contextos celulares muy diversos, como vivir dentro de otras bacterias o formar biofilms resistentes.
Referencias
- Rebecca J. Parr, Yoann G. Santin, Giedrė Ratkevičiūte, et al. A porin-like protein used by bacterial predators defines a wider lipid-trapping superfamily. Nature Communications (2025). DOI: 10.1038/s41467-025-61633-0.
Cortesía de Muy Interesante
Dejanos un comentario: