En el colegio, probablemente alguien mencionó que buena parte del ADN humano es “basura”. Fragmentos sin función aparente, reliquias genéticas inservibles que simplemente estaban ahí, ocupando espacio. Pero la ciencia acaba de dar un vuelco a esa idea. Un grupo internacional de investigadores ha encontrado que parte de ese ADN “basura” es en realidad un código oculto heredado de virus antiguos, que regula cómo se activan nuestros genes más fundamentales.
Este hallazgo, publicado en Science Advances, no solo corrige un error conceptual, sino que revela un mecanismo profundo en la evolución humana y de otros primates. Según el estudio, fragmentos virales integrados en nuestro genoma hace millones de años funcionan como interruptores genéticos que influyen en procesos claves del desarrollo temprano. Y lo más sorprendente: este código viral no está enterrado sin función. Está activo, mutando y posiblemente contribuyendo a lo que nos hace humanos.
El legado viral que llevamos dentro
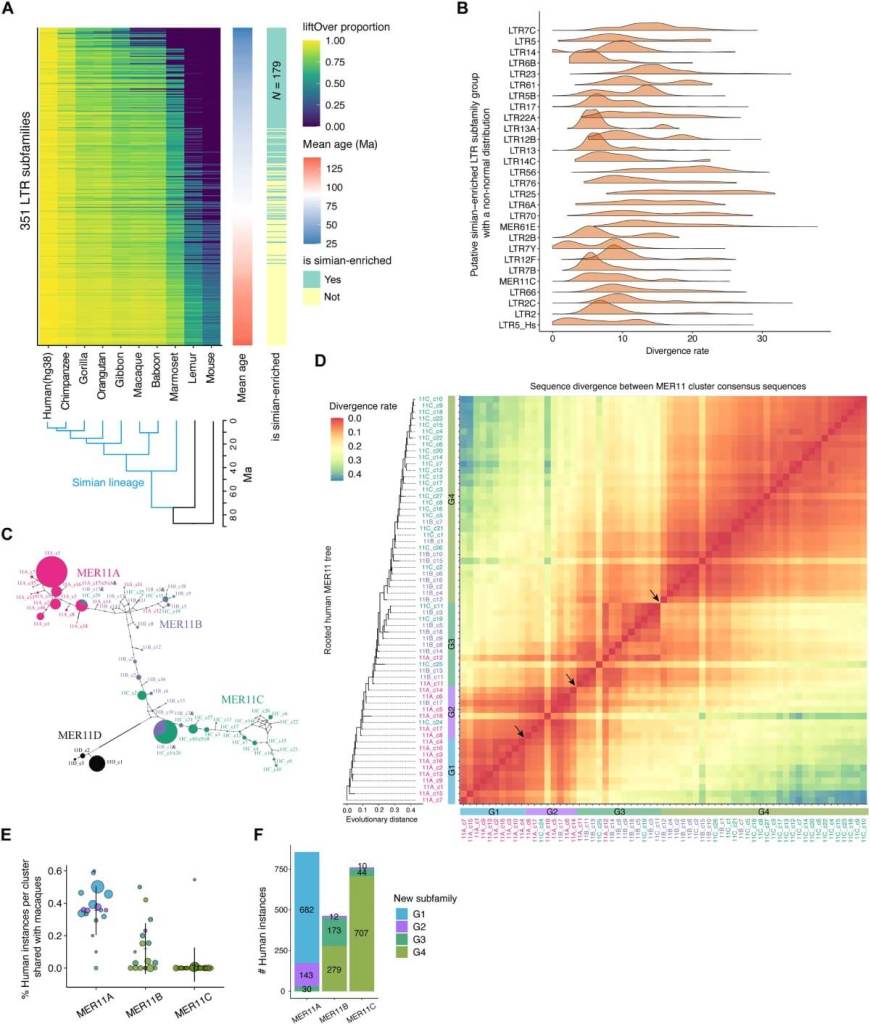
Una parte significativa de nuestro genoma está formada por lo que se conoce como elementos retrovirales endógenos. Estos son restos de antiguos virus que, en lugar de desaparecer tras infectarnos, se integraron en nuestro ADN y se transmitieron de generación en generación. Algunos de estos elementos, como los denominados MER11, se expandieron durante la evolución de los primates y ocupan hoy vastas regiones del genoma humano.
Durante mucho tiempo, estas secuencias fueron ignoradas o clasificadas como irrelevantes. Pero el nuevo estudio demuestra que su organización actual en las bases de datos genómicas era imprecisa y limitada, lo que impedía entender su verdadero papel. Para resolver esto, los científicos desarrollaron un enfoque filogenético que permitió identificar cuatro nuevas subfamilias dentro del grupo MER11, denominadas MER11_G1 a MER11_G4, en orden de antigüedad .
Un método innovador para descifrar el genoma
En lugar de confiar en métodos automáticos de clasificación como RepeatMasker, los investigadores compararon directamente las secuencias MER11 en humanos, chimpancés y macacos. Así detectaron patrones de conservación evolutiva que no se habían reconocido hasta ahora. Esta estrategia reveló que al menos el 30 % de las secuencias estudiadas estaban mal anotadas y debían ser reclasificadas.
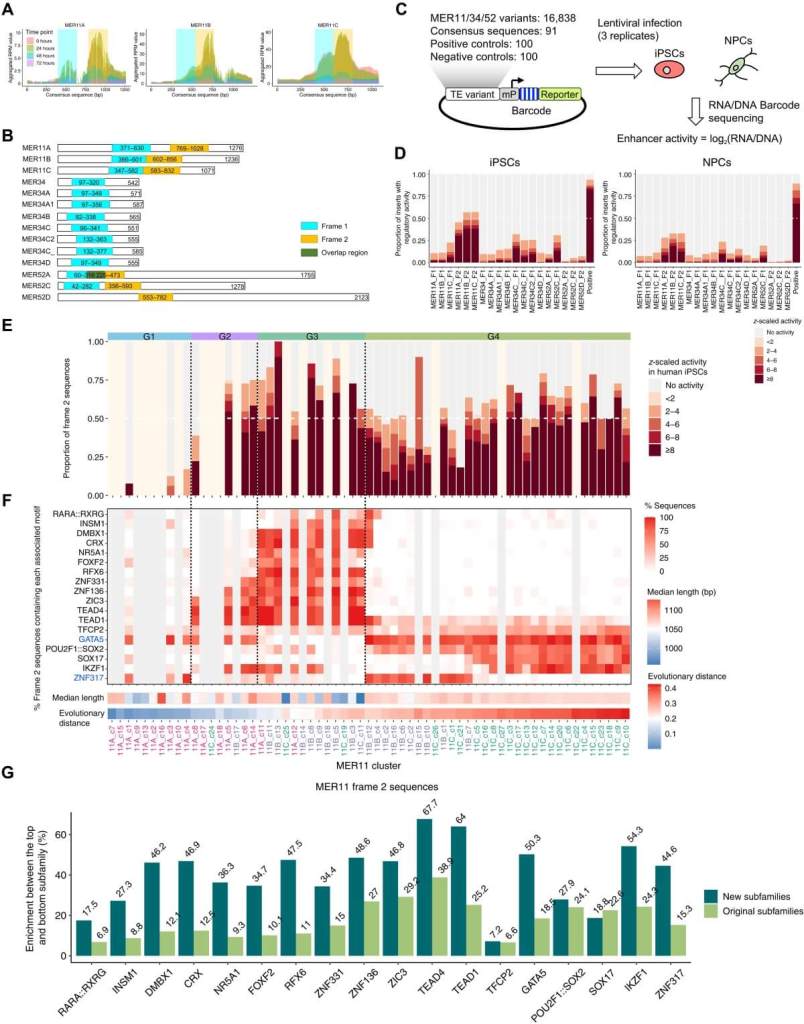
El paso siguiente fue comprobar si estas nuevas subfamilias tenían funciones reales en la regulación de genes. Para ello, el equipo utilizó un método llamado lentiMPRA (ensayo masivo de reporteros por lentivirus). Esta técnica permite probar miles de secuencias de ADN al mismo tiempo, insertándolas en células y midiendo si activan o no la expresión genética.
Los resultados fueron contundentes: la subfamilia más joven, MER11_G4, demostró una gran capacidad para activar genes en células madre humanas, lo que sugiere que no solo está activa, sino que podría estar influyendo en etapas clave del desarrollo embrionario .
Interruptores genéticos heredados de virus
La activación de genes no ocurre al azar. En el núcleo de la célula, factores de transcripción (proteínas especializadas) se unen a secuencias específicas de ADN conocidas como motivos regulatorios. Los investigadores encontraron que MER11_G4 contiene motivos únicos que funcionan como puntos de anclaje para estos factores. En concreto, observaron una acumulación reciente de motivos del tipo SOX y POU::SOX2 en humanos y chimpancés, que no aparecen en los macacos .
Estas diferencias pueden parecer pequeñas, pero tienen implicaciones profundas. Como explica el estudio, una única deleción de nucleótido bastó para que surgiera un nuevo motivo SOX funcional, lo que incrementó la capacidad de esa secuencia para activar genes. Según los autores, esto sugiere que la evolución de estos elementos no solo fue pasiva, sino que fue moldeada activamente por cambios que favorecieron su función reguladora.
La cita literal del paper lo deja claro: “una única deleción de nucleótido condujo a la ganancia de motivos relacionados con SOX, lo que aumentó el potencial regulador de las instancias en esta subfamilia evolutivamente joven”.
Una contribución silenciosa a la evolución
Uno de los aspectos más llamativos del estudio es la forma en que estas secuencias, lejos de ser inertes, han adquirido funciones nuevas a lo largo del tiempo. No solo conservan actividad reguladora, sino que interactúan con elementos epigenéticos, como modificaciones en histonas o marcas químicas que controlan el acceso al ADN. En palabras de los autores, cada subfamilia presenta “perfiles epigenéticos distintos”, lo que refuerza la idea de que desempeñan funciones diferenciadas en distintas etapas del desarrollo .
El análisis también reveló que los perfiles epigenéticos más activos coincidían con las subfamilias más jóvenes, como MER11_G3 y G4. Esto sugiere que estas secuencias siguen evolucionando, y que su papel regulador podría estar aún expandiéndose en nuestra especie. En otras palabras, lo que fue un virus hace millones de años ahora podría estar regulando genes clave en humanos modernos.
Además, las diferencias observadas entre humanos, chimpancés y macacos indican que estos cambios podrían haber contribuido a la aparición de rasgos específicos en cada especie. Si bien aún falta investigación funcional directa, los resultados apuntan a que estos elementos podrían haber influido en los caminos evolutivos que nos separan de otros primates.
Reescribiendo la definición de “ADN basura”
Este estudio representa un avance clave en la comprensión de nuestro genoma. Al redefinir y clasificar con mayor precisión los elementos MER11, los investigadores han abierto la puerta a nuevas formas de explorar cómo se originan y diversifican los elementos reguladores en el ADN.
Lo más relevante es que este trabajo desafía directamente la idea de que el ADN repetitivo y de origen viral no sirve para nada. Por el contrario, demuestra que puede convertirse en parte del engranaje que regula los procesos más esenciales de la vida. Esto obliga a repensar la forma en que entendemos el “ruido” genético y a reconocer que, en muchos casos, ese ruido es música que aún no sabíamos escuchar.
Como afirma el estudio: “nuestro genoma fue secuenciado hace tiempo, pero aún desconocemos la función de muchas de sus partes”. Y este nuevo descubrimiento podría ser el primer paso para entender una de sus partituras más antiguas y fundamentales.
Referencias
- Xun Chen, Zicong Zhang, Yizhi Yan, Clement Goubert, Guillaume Bourque, Fumitaka Inoue. A phylogenetic approach uncovers cryptic endogenous retrovirus subfamilies in the primate lineage. Science Advances, 11(29):eads9164. https://doi.org/10.1126/sciadv.ads9164.
Cortesía de Muy Interesante
Dejanos un comentario: