Una célula del sistema inmunitario no se queda quieta. Es una exploradora incansable que atraviesa tejidos, sigue señales químicas y responde a las amenazas. Esta movilidad es esencial para mantenernos con vida, y sin embargo, los mecanismos que la hacen posible han sido, durante décadas, un misterio complejo incluso para la ciencia. ¿Cómo saben las células a dónde ir? ¿Cómo evitan perderse en el intrincado laberinto del cuerpo humano?
Ya sabemos que las células “oyen” y que generan impulsos eléctricos. Ahora viene el siguiente capítulo en la historia de la biología. En un estudio reciente publicado en Cell, un equipo internacional de investigadores ha descifrado una parte clave de este enigma: han identificado el código molecular que permite a ciertas proteínas guiar con precisión el movimiento celular. Es un avance técnico y conceptual que no solo responde una de las grandes preguntas de la biología, sino que abre la puerta a manipular este proceso con fines médicos. Sí, ahora los científicos pueden modificar la forma en que una célula se mueve, y hacia dónde.
El lenguaje secreto de las células en movimiento
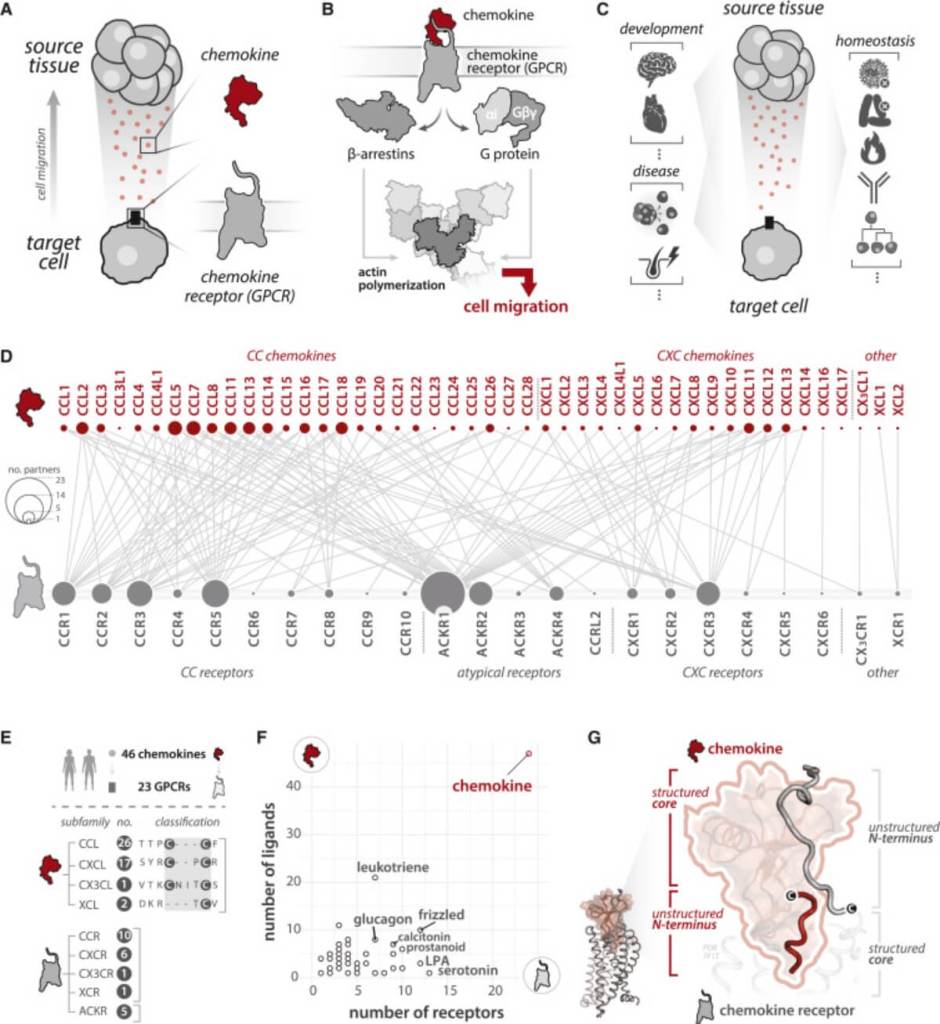
Las células no se mueven al azar. Siguen señales emitidas por otras células que liberan unas moléculas llamadas quimioquinas, que se unen a unos receptores especiales llamados GPCRs (receptores acoplados a proteínas G). Esta pareja —quimioquina y receptor— es la que determina si una célula avanza, gira o se detiene. El problema es que existen 46 quimioquinas y 23 receptores de este tipo en el ser humano, y muchos de ellos son muy similares entre sí. Entender cómo se emparejan correctamente ha sido una tarea extremadamente difícil.
El equipo liderado por Andrew Kleist y M. Madan Babu encontró una solución inesperada. Descubrieron que la selección entre quimioquina y receptor no depende solo de su forma estructural, sino también de regiones desordenadas en sus secuencias. Estas partes “caóticas” de la proteína, lejos de ser inútiles, contienen información esencial para la identificación mutua. Como indican los autores: “La información para cómo las quimioquinas y los GPCRs se seleccionan entre sí está almacenada en pequeños paquetes discretos de regiones altamente desordenadas”.
Claves públicas y privadas: una analogía digital hecha realidad biológica
Para explicar este sistema, los investigadores recurrieron a una comparación con la criptografía digital. En una transacción segura por internet, los participantes usan claves públicas y privadas para garantizar la seguridad de la comunicación. De manera análoga, las quimioquinas y los receptores usan combinaciones de regiones estructuradas (clave pública) y desordenadas (clave privada) para reconocerse de forma exclusiva.
Este método garantiza que una célula responda únicamente a la señal adecuada, sin interferencias. Las regiones desordenadas actúan como “firmas químicas únicas” que aseguran que el emparejamiento sea correcto. Esta organización no es una excepción, sino una regla general: “La interacción de una región desordenada de la quimioquina con una región estructurada del receptor proporciona a las células un identificador químico único”. Esta clave es lo que desencadena, finalmente, el movimiento.
Diseñando el tráfico celular a medida
Con esta información en la mano, el siguiente paso fue demostrar que se podía modificar. Y lo lograron. Los investigadores alteraron regiones específicas de una quimioquina y comprobaron que podían cambiar su capacidad para unirse a distintos receptores. En un experimento especialmente significativo, lograron modificar el movimiento de linfocitos T, un tipo de glóbulo blanco esencial para el sistema inmune, bloqueando una señal que normalmente los detiene.
Esto no es solo una curiosidad de laboratorio. Significa que podríamos redirigir células inmunitarias para que ataquen tumores, o guiar células madre a los lugares donde se necesiten. Como dijo Babu: “Con este entendimiento, ahora podemos introducir pequeños cambios en la estructura de una quimioquina para alterar el movimiento celular de forma dirigida”.

Un mapa evolutivo y personalizado del movimiento celular
El estudio también reveló que muchas de estas “claves” están distribuidas de forma jerárquica. Algunas son conservadas, compartidas entre muchos tipos de células y especies; otras son específicas de subfamilia, y unas terceras son totalmente individualizadas. Esta arquitectura permite a las células disponer de un sistema flexible, adaptativo y preciso.
Los investigadores utilizaron más de 1.000 secuencias de proteínas de distintos organismos y más de una docena de estructuras moleculares tridimensionales para identificar estas diferencias. Observaron que la mayoría de las interacciones críticas ocurren en regiones sin una estructura fija, especialmente en los extremos de las proteínas. Estas regiones albergan lo que los científicos llaman SLiMs (Short Linear Motifs), pequeños fragmentos que funcionan como interruptores funcionales clave.
Aplicaciones médicas: más allá de la teoría
La utilidad de estos hallazgos no es meramente académica. En el propio estudio, los autores aplicaron sus principios a una proteína viral llamada vMIP-II, utilizada por un herpesvirus para evadir el sistema inmune. Al modificar tres residuos clave, lograron que esta proteína perdiera afinidad por ciertos receptores humanos y ganara eficacia en otros.
Esta manipulación dirigida demuestra que es posible rediseñar proteínas naturales para cambiar su comportamiento, una idea poderosa con aplicaciones potenciales en inmunoterapia, enfermedades autoinmunes, trasplantes y más. Tal como afirma el artículo: “Los principios básicos de selectividad y promiscuidad identificados pueden guiar cambios específicos en las secuencias de quimioquinas para modular las preferencias de unión y sus efectos sobre el movimiento celular”.

La promesa de un tráfico celular inteligente
Uno de los problemas de las terapias celulares, como las CAR-T, es que no siempre llegan al sitio adecuado o lo hacen de forma ineficiente. Modificar la forma en que estas células navegan por el cuerpo puede ser la clave para mejorar su eficacia. Los autores proponen que los receptores quimioquínicos y sus ligandos pueden actuar como puertas lógicas programables, capaces de tomar decisiones en función del entorno celular.
Este enfoque podría dar lugar a tratamientos más seguros y personalizados, en los que las células se activen solo cuando estén en el sitio correcto, evitando efectos secundarios indeseados. Todo esto, partiendo del conocimiento de un sistema que parecía caótico, pero que ahora entendemos como un código perfectamente organizado y manipulable.
Referencias
- Kleist, A.B., Szpakowska, M., Talbot, L.J., et al. (2025). Encoding and decoding selectivity and promiscuity in the human chemokine-GPCR interaction network. Cell, 188, 1–20. https://doi.org/10.1016/j.cell.2025.03.046.
Cortesía de Muy Interesante
Dejanos un comentario: