El hallazgo de un individuo prehispánico en El Saucillo, Zimapán (Hidalgo, México) ha permitido reconstruir un fragmento de la historia invisible de las antiguas poblaciones mesoamericanas: la de sus microorganismos intestinales. El estudio del microbioma intestinal de este hombre, fallecido hace unos 936 años, revela un ecosistema bacteriano sorprendentemente complejo cuyo rastro se ha conservado tanto en los restos del tejido intestinal momificado como en las paleoheces. El análisis, publicado en 2025 por un equipo multidisciplinario de la Universidad Nacional Autónoma de México (UNAM) y del Instituto Nacional de Antropología e Historia (INAH), ofrece la primera caracterización del microbioma intestinal de un individuo del México prehispánico.
El individuo de Zimapán y su contexto arqueológico
Los restos humanos fueron descubiertos en 2014 por habitantes locales, quienes los entregaron al Instituto Nacional de Antropología e Historia (INAH). El cuerpo, que estaba envuelto en un fardo funerario, se hallaba en un abrigo rocoso. Las condiciones microambientales de este emplazamiento favorecieron una conservación excepcional de los tejidos blandos, incluyendo la piel, los vasos sanguíneos, los fragmentos intestinales y la materia fecal.
Las dataciones por radiocarbono situaron al individuo —un varón joven de entre 21 y 35 años— hacia el Periodo Posclásico temprano (900–1200 d.C.), un momento de intensa interacción cultural entre Mesoamérica y Aridoamérica. La ubicación fronteriza del hallazgo ha permitido asociarlo con la cultura otopame, una de las más antiguas del altiplano central. El cuerpo del individuo se depositó sobre una capa orgánica compuesta por pastos y vegetación xerófila, propios de un entorno semidesértico que condicionó su dieta y modo de vida.
De la paleoheces al ADN bacteriano
El grupo de investigación procedió a extraer muestras de ADN antiguo del cuerpo. Así, se analizaron dos muestras de paleoheces y una de tejido intestinal momificado. Mediante la secuenciación masiva del gen 16S rRNA, se logró recuperar entre 200 y 400 ng/μl de ADN, con más de un millón de lecturas válidas en total.
Los datos se procesaron con el software QIIME2 y la base de datos SILVA 138, utilizando algoritmos de clasificación taxonómica y análisis multivariante. Para comparar la composición bacteriana, el equipo integró secuencias de microbiomas modernos —humanos, de suelo y compost— y antiguos, como los procedentes de las momias andinas y los coprolitos medievales europeos.

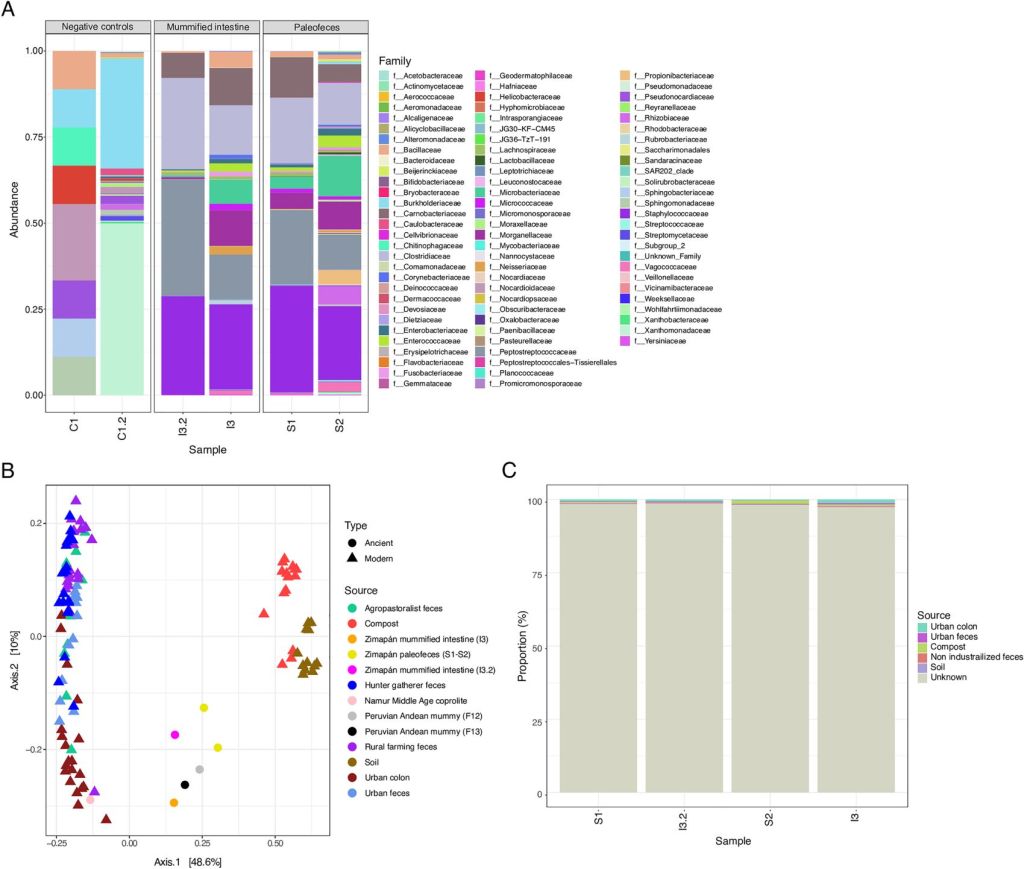
Resultados: un ecosistema intestinal antiguo
Los resultados revelaron que las familias bacterianas más abundantes presentes en el indiciduo fueron las Peptostreptococcaceae, Clostridiaceae, Enterobacteriaceae, Enterococcaceae y Lachnospiraceae, todas ellas componentes “habitantes” habituales del intestino humano. En contraste, los controles negativos mostraron perfiles completamente distintos, lo que confirma la autenticidad de los datos.
Los científicos localizaron la Romboutsia hominis, un integrante exclusivo del intestino humano moderno que, hasta el momento, no se había identificado en microbiomas antiguos. Asimismo, el género Clostridium apareció en gran abundancia, tal y como sucede en las momias precolombinas de los Andes. Estos microorganismos, fermentadores de polisacáridos complejos, producen ácidos grasos de cadena corta esenciales para la homeostasis intestinal. Su presencia, por tanto, podría reflejar una dieta rica en fibras vegetales e insectos, coherente con la tradición entomófaga documentada en el actual estado de Hidalgo.
La familia Lachnospiraceae, por otro lado, también se detectó de forma abundante. Estas bacterias son clave en la degradación de celulosa y hemicelulosa, polisacáridos vegetales imposibles de digerir por el ser humano sin ayuda bacteriana. La prevalencia de estos taxones sugiere una alimentación diversa basada en recursos del ecosistema xerófilo, como agaves, yucas o nopales.

Comparaciones y análisis multivariante
El equipo de estudio aplicó un análisis de coordenadas principales (PCoA) para comparar las muestras de Zimapán con otros microbiomas procedentes de distintos entornos. Los resultados mostraron que los datos mexicanos se acercan a los proporcionados por las momias andinas precolombinas, así como al microbioma intestinal moderno. Esta proximidad sugiere una continuidad funcional entre los microbiomas prehispánicos de América, a pesar de sus diferencias geográficas.
Con todo, las muestras de Zimapán también presentaron una ligera desviación respecto al microbioma humano moderno, un dato que se ha atribuido a la presencia de bacterias ambientales como Jeotgalicoccus o Sphingomonadaceae. Estos grupos, comunes en suelos y sedimentos, probablemente se introdujeron de forma secundaria durante la descomposición post mortem. El análisis SourceTracker2, por otro lado, confirmó que más del 90 % del material genético no se corresponde con fuentes modernas conocidas, lo que indica la singularidad del ecosistema bacteriano antiguo.
Microbioma, dieta y entorno: una interpretación biocultural
El hombre de Zimapán ha permitido explorar la vida en el México prehispánico desde un punto de vista radicalmente distinto. El predominio de bacterias fermentadoras y degradadoras de polisacáridos vegetales apunta a una dieta rica en fibras naturales, procedentes, con probabilidad, de plantas silvestres y cultivos locales. La posible presencia de enzimas asociadas a la degradación de quitina, ya observadas en estudios similares, refuerza la hipótesis del consumo de insectos.
El entorno semidesértico del Valle del Mezquital y las prácticas agrícolas tradicionales otopames sustentan esta interpretación. Así, los microorganismos presentes en el intestino, al conservar señales bioquímicas de los hábitos alimentarios, la salud intestinal y las adaptaciones ecológicas, se convierten en testigos microscópicos de las estrategias de subsistencia prehispánicas.

Limitaciones y perspectivas
Pese a los logros, los investigadores subrayan que algunas familias bacterianas comunes en microbiomas humanos —como Bacteroidaceae, Prevotellaceae o Ruminococcaceae— no se detectaron o aparecieron en baja abundancia. Esto podría deberse a la fragmentación del ADN antiguo, característico de muestras de más de un milenio de antigüedad.
Para superar esas limitaciones, el equipo propone realizar investigaciones en el futuro que se basen en la metagenómica, una técnica capaz de reconstruir genomas bacterianos completos y evaluar los patrones de daño del ADN. Ello permitiría confirmar la autenticidad de los microorganismos detectados y explorar sus funciones metabólicas en detalle.
Este primer estudio, sin embargo, marca un hito en la paleomicrobiología mexicana, al demostrar la viabilidad de recuperar información genética de microbiomas prehispánicos para poder compararlos con referencias globales. Así, el microbioma del hombre de Zimapán constituye un testimonio microscópico de la vida cotidiana en el México anterior a la colonización, capaz de ofrecer una perspectiva inédita sobre la evolución biocultural de nuestra especie en el continente americano.
Referencias
- Rosas-Plaza, S. et al. 2025. “Microbiome characterization of a pre-Hispanic man from Zimapán, Mexico: Insights into ancient gut microbial communities”. PLOS ONE, 20(10): e0331137. DOI: https://doi.org/10.1371/journal.pone.0331137
Cortesía de Muy Interesante
Dejanos un comentario: