La biofísica es una ciencia que estudia la biología desde el punto de vista y con los métodos de la física para entender cómo funcionan los sistemas biológicos. Es una de las primeras ciencias que conlleva un enfoque interdisciplinar, combinando la física, la química y la biología para estudiar los seres vivos.
Cuando se incorpora la informática a este enfoque interdisciplinar, nace la biofísica computacional, una rama que permite simular y modelar matemáticamente sistemas biológicos con gran precisión. Además, la utilización de herramientas computacionales nos permite el estudio de los seres vivos a niveles macroscópico, molecular o cuántico, una escala y nivel de detalle inalcanzables con un estudio meramente experimental.
Por tanto, la biofísica computacional facilita la creación de modelos que describen interacciones químicas y dinámicas en sistemas biológicos, permitiendo predecir comportamientos moleculares. Esta disciplina tiene unas áreas de investigación básicas, como son la dinámica molecular, para estudiar el movimiento, plegamiento o interacción de las proteínas, el modelado de membranas biológicas, con el estudio de estructura y función de las membranas celulares, o el estudio de redes bioquímicas y metabólicas, incluyendo las redes de señalización celular.
Así pues, la biofísica computacional requiere la obtención, el estudio y la integración de una gran cantidad de datos. Sin embargo, interpretar este volumen de datos de forma eficiente supera las capacidades humanas o los métodos tradicionales. Precisamente, aquí es donde entra en juego la inteligencia artificial (IA), una herramienta cada vez más presente en este campo.
Gracias a su capacidad para detectar patrones complejos y aprender de ejemplos previos, la IA se ha convertido en una aliada poderosa para acelerar descubrimientos y extraer conocimiento de datos experimentales y computacionales. En consecuencia, su integración en biofísica computacional representa un avance prometedor para comprender mejor el funcionamiento de los sistemas biológicos a nivel molecular.
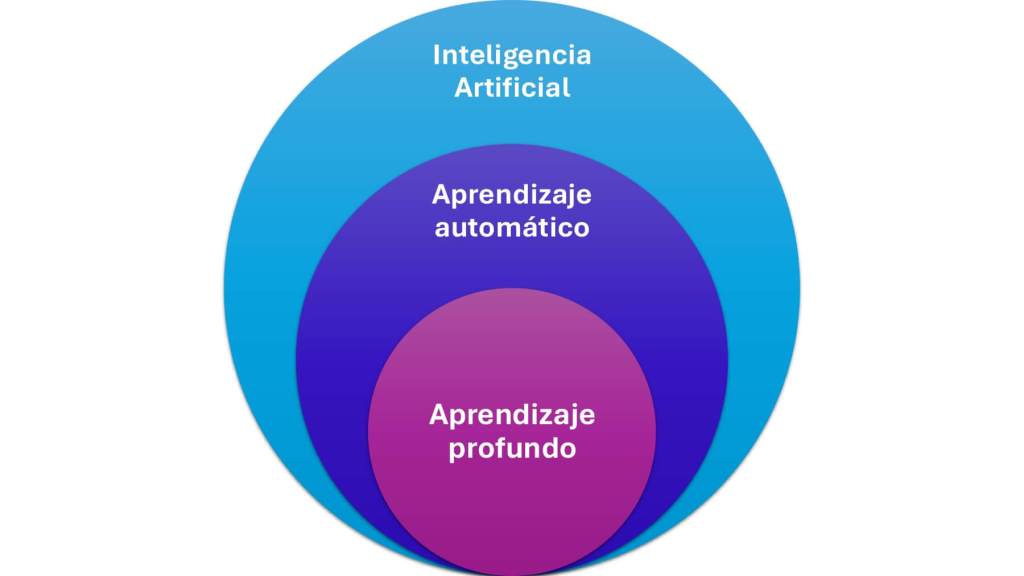
¿Qué tipos de IA se usan en biofísica computacional?
La IA es una rama de la informática que busca realizar y automatizar tareas que normalmente requieren inteligencia humana, como reconocer imágenes, entender el lenguaje, tomar decisiones o aprender de la experiencia. En cualquier caso, la IA requiere de una gran cantidad de datos y suficiente poder computacional para poder entrenar los modelos y reconocer patrones, hacer predicciones o incluso generar soluciones nuevas.
La IA se divide en varias ramas, pero las más utilizadas en ciencia son el aprendizaje automático (machine learning) y el aprendizaje profundo (deep learning). En primer lugar, el aprendizaje automático es una división dentro de la IA que permite a los algoritmos aprender de grandes volúmenes de datos sin ser programados explícitamente, encontrando patrones y realizando predicciones sobre datos nuevos. Por ejemplo, el aprendizaje automático se puede utilizar para clasificar el tipo de estructuras que adopta una proteína en función de parámetros biofísicos. El modelo no es capaz de memorizar, solo detecta patrones subyacentes en nuestros datos.
En segundo lugar, el aprendizaje profundo, una subdivisión dentro del aprendizaje automático, emplea redes neuronales artificiales profundas, inspiradas en el funcionamiento del cerebro humano. Una red neuronal es un modelo matemático compuesto por una capa (red neuronal simple) o por varias (red neuronal profunda). Así, una red neuronal profunda permite que cada capa procese la información y la transmita a la siguiente, alcanzando el reconocimiento de patrones más complejos. Por tanto, el aprendizaje profundo es más potente que el aprendizaje automático y se puede usar, por ejemplo, para reconocer, contar y analizar células a partir de una imagen.
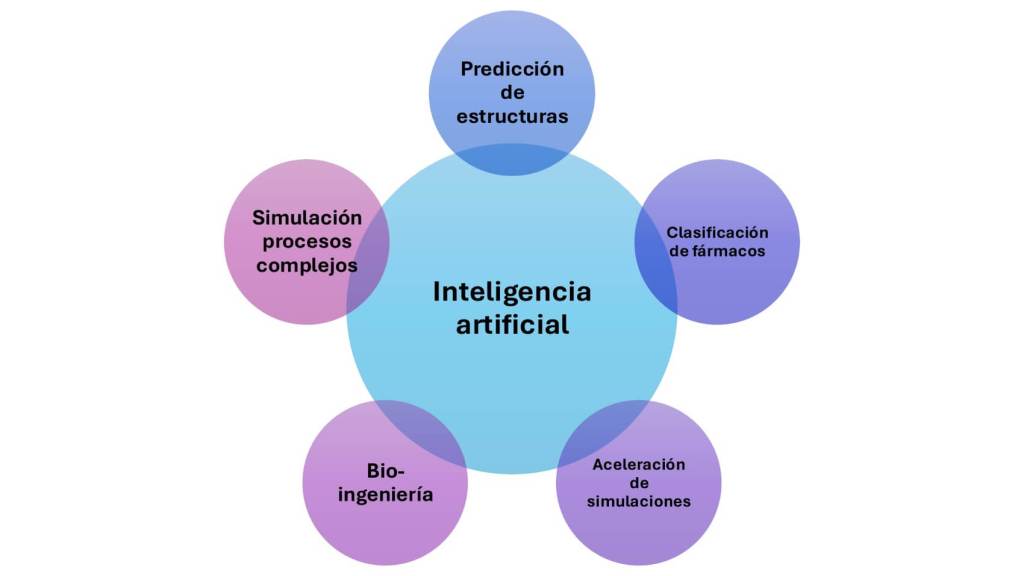
¿Qué puede hacer la IA por la biofísica computacional? Aplicaciones concretas y casos de éxito
El aprendizaje automático y el aprendizaje profundo se están utilizando cada vez más comúnmente en el campo de la biofísica computacional, permitiendo un avance significativo en varias áreas de la biofísica. Por ejemplo, la IA ha mejorado la predicción de estructuras moleculares, el proceso de diseño de fármacos y biomoléculas, el análisis automático y la aceleración de simulaciones moleculares o la simulación de procesos complejos.
Predicción de estructuras moleculares
Las proteínas están formadas por una secuencia de aminoácidos, lo que les confiere una capacidad de plegarse y adoptar una estructura tridimensional. Normalmente, dicha estructura 3D está estrechamente relacionada con la función que realiza la proteína, ergo la determinación de la estructura tridimensional de una molécula (principalmente las proteínas) es clave para entender enfermedades, diseñar fármacos o modificar funciones biológicas.
Esta tarea se realizaba comúnmente con técnicas experimentales, como la cristalografía de rayos X, la resonancia magnética nuclear (RMN) o la criomicroscopía electrónica, que son procesos lentos y costosos. No obstante, todo cambió con la llegada de la IA. En el año 2020, el modelo AlphaFold, desarrollado por DeepMind, cambió las reglas del juego al demostrar que una IA podía predecir la estructura tridimensional de las proteínas solo a partir de su secuencia, precisamente, gracias a una red neuronal basada en el aprendizaje profundo.
AlphaFold ha conseguido tal hito gracias al estudio de homologías de secuencia y al estudio de la coevolución de secuencias, es decir, qué secuencias y mutaciones (cambios en las secuencias) han evolucionado junto con otras. Esto permite la predicción de la estructura tridimensional con base en otras proteínas con estructura 3D ya conocida. La importancia de este modelo se demostró el año pasado, dado que se otorgó el premio Nobel a los líderes del proyecto AlphaFold (Demis Hassabis y John M. Jumper) junto con el creador de Rosetta (David Baker), un modelo que se puede utilizar para crear proteínas desde cero.
Clasificación de fármacos y biomoléculas
Una de las estrategias utilizadas en el proceso de diseño de fármacos consiste en la identificación de nuevas moléculas que se unan con alta afinidad y precisión a una proteína de interés. El primer paso es el análisis informático de una gran cantidad de moléculas, que se puede realizar a gran escala gracias a la potencia computacional. Tras esta etapa de cribado virtual, se seleccionan las moléculas más prometedoras, que luego se sintetizan y evalúan experimentalmente. Este enfoque permite explorar un amplio espacio químico de manera eficiente, reduciendo significativamente el tiempo y los costos asociados al ensayo experimental de cada candidato.
Gracias a la IA, se pueden identificar compuestos terapéuticos todavía más rápida y precisamente, con una mejor relación coste-efectividad, abriendo nuevas posibilidades en la medicina personalizada y el tratamiento de enfermedades complejas. Por ejemplo, GENNIUS es un modelo de aprendizaje profundo que permite predecir las interacciones proteína-fármaco más precisas.
Análisis automático y aceleración de simulaciones moleculares
El uso de la IA en el campo de las simulaciones moleculares representa una de las áreas más prometedoras de la biofísica computacional, pues permite hacer simulaciones más rápidas y precisas. En primer lugar, las simulaciones moleculares son modelos computacionales que permiten estudiar el comportamiento y movimiento de átomos y moléculas. Se pueden utilizar para ver la interacción de moléculas a nivel atómico, explorar transiciones estructurales o estudiar la estructura y dinamismo de las membranas celulares.
Así pues, los aprendizajes automático y profundo permiten calcular la energía de los sistemas moleculares con mucha mayor precisión y menos tiempo, analizar las grandes cantidades de información generadas por las simulaciones, o incluso generar modelos de predicción y clasificación de las proteínas de las cuales no se tiene información más allá de la secuencia. Por ejemplo, SchNet o DeePMD son algoritmos de aprendizaje profundo que permiten una caracterización energética y a nivel atómico de las proteínas.
Rediseño de biomoléculas y bioingeniería
La IA también ha abierto nuevas fronteras en el diseño de biomoléculas y la bioingeniería, permitiendo crear proteínas o sistemas celulares con funciones mejoradas o incluso desde cero. Por ejemplo, en lugar de partir de la secuencia, se puede diseñar una estructura con una función determinada y crear una secuencia que realice dicha función. Además, también permite crear nuevas moléculas de proteína o ADN que no existen en la naturaleza.
Existen varios modelos que utilizan aprendizaje automático y aprendizaje profundo para generar estas estructuras, como por ejemplo RFDiffusion, o también Rosetta y AlphaFold. Estos modelos permiten generar estructuras estables con facilidad, diseñar enzimas con funciones específicas o incluso se han utilizado para acelerar el diseño de vacunas, entre otros, del virus SARS-CoV-2.
Simulación de procesos biológicos complejos
Uno de los campos más complicados de analizar y predecir es la simulación de procesos biológicos complejos, dado que involucran múltiples componentes interactuando entre sí. Por ejemplo, las redes de señalización celular, que implican varias proteínas, la regulación génica, que requiere el uso de varias moléculas, o la interacción célula-entorno, que comprende el estudio de una célula en conjunto.
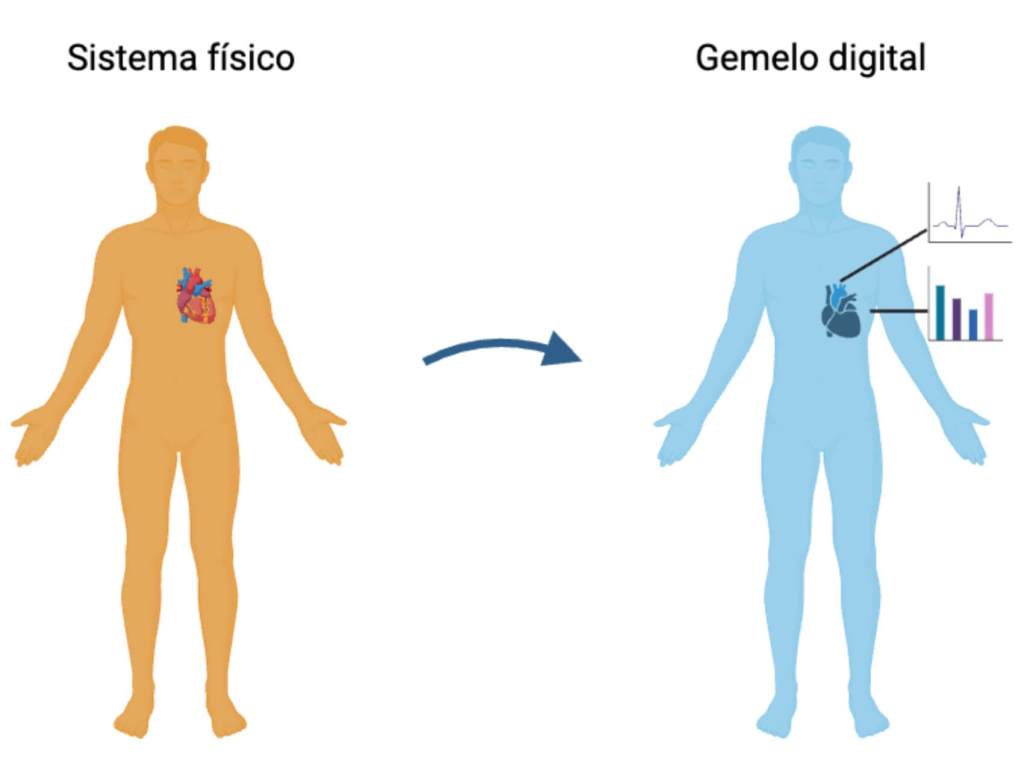
Así pues, la IA permite predecir, extrapolar y simplificar el estudio de sistemas complejos, ya que permite generar modelos matemáticos que simulen dichos procesos. En este sentido, se pueden generar modelos de IA que aprendan el comportamiento de las redes o procesos de regulación, se puede predecir la respuesta inmunitaria a un patógeno o incluso se ha llegado a generar simulaciones de sistemas enteros, conocidos como digital twins (gemelos digitales).
Precisamente, un gemelo digital es un modelo matemático avanzado que se genera a partir de la recopilación masiva de datos procedentes de sensores, imágenes médicas, análisis clínicos y otras fuentes. Estos modelos replican con gran fidelidad el comportamiento del organismo original, lo que permite predecir cómo respondería ante distintas condiciones o intervenciones.
Una de las grandes ventajas de los gemelos digitales es que permiten simular distintos tratamientos de forma simultánea para identificar el más eficaz antes de aplicarlo. Esto ayuda a reducir riesgos, personalizar terapias y optimizar recursos. Además, facilitan el seguimiento de enfermedades y mejoran la toma de decisiones clínicas, abriendo paso a una medicina más precisa y centrada en el paciente.
La IA ofrece mejoras respecto al campo de la biofísica computacional. Entre sus principales ventajas destacan su rapidez, la capacidad de explorar una amplia variedad de estructuras y moléculas, y la posibilidad de reducir el número de experimentos fallidos al predecir resultados mediante modelos computacionales. Aparte, permite simular condiciones experimentales que serían difíciles de reproducir en un laboratorio.
Además, la IA ofrece la posibilidad de descubrir patrones en datos experimentales, automatizar procesos y ayuda a personalizar tratamientos, en pos de llegar a un tratamiento de medicina personalizada. Otra ventaja de la IA es su capacidad para proponer soluciones no convencionales que no estarían limitadas por la intuición o experiencia humanas. Al no estar sujeta a las mismas restricciones cognitivas que los humanos, puede detectar patrones ocultos y explorar enfoques innovadores que podrían pasar desapercibidos para las personas.
Sin embargo, la integración de la IA en la biofísica computacional no está exenta de desafíos. En primer lugar, los modelos de IA dependen de la cantidad y calidad de los datos, dado que los sesgos o errores pueden traducirse en predicciones incorrectas. Además, también existe el reto de interpretar los modelos de IA, pues en algunos casos es difícil entender el proceso que sigue el modelo para tomar decisiones. Por último, hay que tener en cuenta que los algoritmos de IA son modelos y, por tanto, hay que comprobar que sus resultados son correctos, lo que hay que hacer experimentalmente.
En paralelo, es importante señalar que el uso de la IA conlleva un considerable impacto ambiental. Su funcionamiento depende de infraestructuras que requieren grandes cantidades de energía, tanto para alimentar los servidores como para refrigerarlos. Por tanto, esta tecnología está lejos de ser una opción verdaderamente sostenible o respetuosa con el medio ambiente.
En resumen, la incorporación de la inteligencia artificial ha revolucionado la biofísica computacional, representando una de las transformaciones más significativas en la forma en que abordamos el estudio de los sistemas biológicos. Gracias a su capacidad de analizar grandes volúmenes de datos, detectar patrones complejos y generar modelos predictivos con una precisión previamente inalcanzable ha ampliado enormemente las posibilidades de investigación en este campo.
No obstante, para que esta revolución alcance todo su potencial, es necesario seguir promoviendo enfoques multidisciplinares, garantizar la calidad de los datos y desarrollar modelos explicables y validarlos experimentalmente. La combinación entre inteligencia humana, inteligencia artificial y experimentación rigurosa es clave para comprender la complejidad de la vida desde una nueva perspectiva computacional.
Referencias
- Jou Mirabent, D., Llebot Rabagliati, J. E., & Pérez García, C. (2008). Física para ciencias de la vida.
- Rodríguez, P. (2018) Inteligencia artificial: Cómo cambiará El Mundo (Y tu vida)
- Jumper, J. et al. (2021) ‘Highly accurate protein structure prediction with alphafold’, Nature, 596(7873), pp. 583–589. doi:10.1038/s41586-021-03819-2.
- Baek, M. et al. (2021) ‘Accurate prediction of protein structures and interactions using a three-track neural network’, Science, 373(6557), pp. 871–876. doi:10.1126/science.abj8754.
- Veleiro, U. et al. (2023) ‘GENNIUS: An ultrafast drug–target interaction inference method based on Graph Neural Networks’, Bioinformatics, 40(1). doi:10.1093/bioinformatics/btad774.
- Schütt, K.T. et al. (2018) ‘SchNet – a deep learning architecture for molecules and materials’, The Journal of Chemical Physics, 148(24). doi:10.1063/1.5019779.
- Wang, H. et al. (2018) ‘DeePMD-kit: A deep learning package for many-body potential energy representation and molecular dynamics’, Computer Physics Communications, 228, pp. 178–184. doi:10.1016/j.cpc.2018.03.016.

Alex Perálvarez-Marín
Doctor en Bioquímica y Biología Molecular


Eric Catalina-Hernández
Estudiante de Doctorado en Bioinformática

Cortesía de Muy Interesante
Dejanos un comentario:
