Nuestro genoma es el conjunto de todo el ADN que tienen nuestras células, el libro de instrucciones para todas las actividades que lleva a cabo nuestro organismo. Más allá del genoma humano, que nos proporciona esta secuencia completa de ADN, otra de las ramas de la bioquímica, la proteómica, nos permite explorar la funcionalidad de las células.
La proteómica consiste en el estudio del conjunto de las proteínas —las encargadas de llevar a cabo la función que definen los genes— expresadas por un organismo en unas condiciones y un momento temporal determinados. Esta herramienta ha revolucionado, desde la obtención del genoma humano completo, la búsqueda de biomarcadores, parámetros que nos permiten conocer el estado de un proceso biológico, por ejemplo, de una enfermedad. Los biomarcadores funcionan como indicadores moleculares medibles y específicos, y nos permitirían diagnosticar patologías de forma temprana, monitorizar su evolución o diseñar tratamientos personalizados.
¿Cómo puede la proteómica aportar nuevas respuestas?
Las técnicas proteómicas, como la espectrometría de masas, nos permiten analizar el contenido proteico completo de una muestra biológica, por ejemplo, cualquier tejido o cualquier fluido biológico, como plasma o suero de la sangre. Gracias a ello, podemos analizar miles de proteínas al mismo tiempo. Este análisis nos permite detectar cambios en los niveles de expresión de proteínas específicas, lo cual facilita la identificación de proteínas asociadas a enfermedades crónicas, incluso en sus etapas más iniciales. Además, si el análisis se hace sobre fluidos biológicos de fácil acceso, como es la sangre, las proteínas identificadas como biomarcadores permitirían la detección de una enfermedad de forma mínimamente invasiva, rápida y a un precio reducido.
Estas técnicas han sido clave para la identificación de biomarcadores en algunas enfermedades crónicas, como el cáncer colorrectal, el glaucoma, o la enfermedad de Alzheimer. Estudios previos de nuestro grupo de investigación, que forma parte de la Unidad Funcional de Investigación de Enfermedades Crónicas (UFIEC) del Instituto de Salud Carlos III (ISCIII), han revelado proteínas en sangre que no deberían estar presentes en condiciones normales, pero que ante una patología se liberan a la sangre. Por tanto, su detección nos llevaría a un diagnóstico temprano de la enfermedad.

Este es el caso de las proteínas TXNDC17 y SLC8A1, cuya presencia en sangre se ha asociado a cáncer colorrectal, principalmente en estadios iniciales de la enfermedad. Otro ejemplo sería el caso de las proteínas HECTD1 y SLC12A5, cuyos altos niveles en sangre se han asociado a la enfermedad de Alzheimer.
Además de la sangre, otro fluido biológico para el diagnóstico de enfermedades crónicas es el humor acuoso, presente en el ojo y de acceso fácil durante las operaciones de cataratas, y en el cual la presencia de altos niveles de las proteínas RAD23B y GAS6 se han asociado a glaucoma. Gracias a ello, en este último caso el análisis de los niveles de dichas proteínas en el humor acuoso obtenido durante las operaciones de cataratas podría permitir detectar predisposición a glaucoma o inicio de la enfermedad para su tratamiento temprano.
Pero, ¿cómo funciona la espectrometría de masas?
Imagina un detective buscando pistas en la escena de un crimen (Figura 1). La espectrometría de masas actúa de forma similar: identifica proteínas en distintas muestras de tejido o fluidos biológicos (escena del crimen) en busca de aquellas que presenten un patrón de expresión diferente entre una muestra de una persona sana y una muestra de una persona enferma. Para ello, todos los objetos de la muestra (proteínas), que podrían ser pistas del crimen, se rompen para transformarlos en objetos más fáciles de investigar (péptidos), de modo que el detective (el espectrómetro de masas) es capaz de interpretarlas fácilmente y sacar conclusiones sobre quien es cómplice o responsable del crimen (proceso biológico que pueda derivar en enfermedad, por ejemplo).
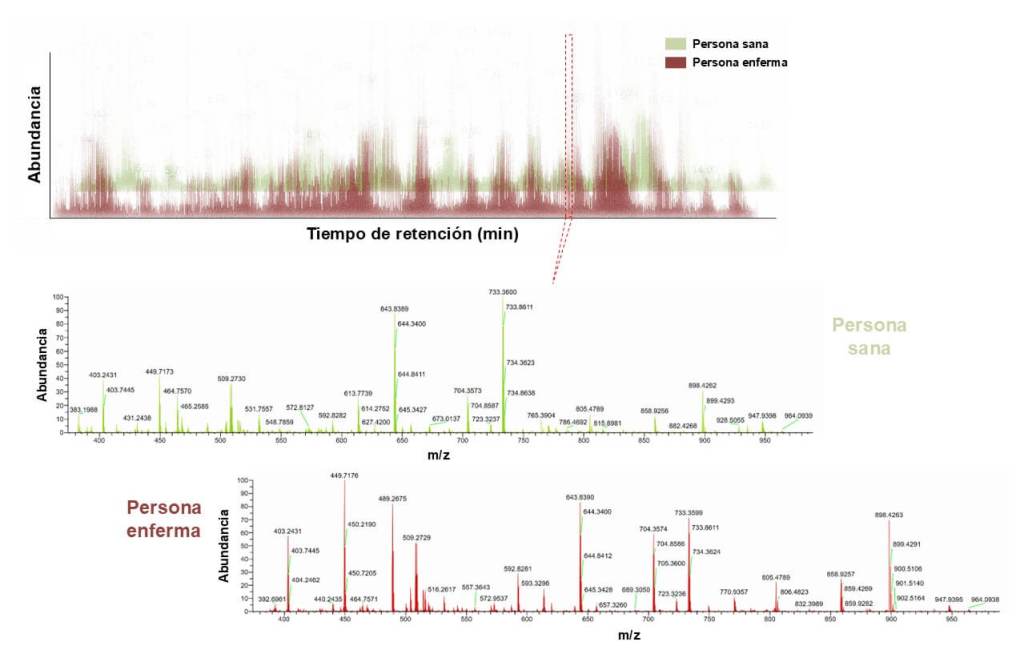
Estas conclusiones las saca el detective a partir del espectro de masas, que sería su mapa de pistas, de manera que en el espectro de masas se encuentra toda la información de todas las pistas de la escena del crimen recogidas por el investigador. Para descubrir al cómplice o responsable del crimen se tienen que analizar todas las pistas, lo cual se hace interpretando el espectro de masas, que ofrece información en forma de gráfico y muestra cómo se distribuyen las pistas según su masa y su carga.
Cada pico en el gráfico representa una pista (péptido). Su altura en el gráfico nos indica cuánto hay de él, y su posición revela su ‘identidad’. Si una de estas pistas aparece en la muestra de una persona enferma, pero no en la muestra de la persona sana, o si en la muestra de la persona sana aparece en menor o mayor intensidad que en la muestra de la persona enferma, ¡es una señal de alerta! Cada pico sería una ‘huella dactilar’ única que el detective detecta, analiza y saca conclusiones (Figura 2). De esta forma se puede obtener una firma compuesta por varias ‘huellas’, que nos indique si hay enfermedad o no en cada muestra.
¿Qué ventajas tiene la proteómica?
La proteómica tiene tres ‘superpoderes’ para ‘cazar’ biomarcadores:
1. Precisión: el microscopio molecular
La proteómica detecta cambios mínimos en las proteínas, como el detective que encuentra una huella dactilar invisible. Esto permite identificar enfermedades en etapas tan tempranas que ni los síntomas han aparecido.
2. Mínimamente invasivo: la prueba del futuro
La proteómica puede analizar fluidos biológicos accesibles como sangre, orina, leche, o humor acuoso, sin necesidad de biopsias o cirugías, lo que permitiría detectar la presencia o ausencia de una enfermedad de forma mínimamente invasiva, como si fuera un análisis de sangre rutinario. Esto reduce riesgos y hace los exámenes más cómodos para los pacientes.
3. Enfoque sistemático: el rompecabezas completo
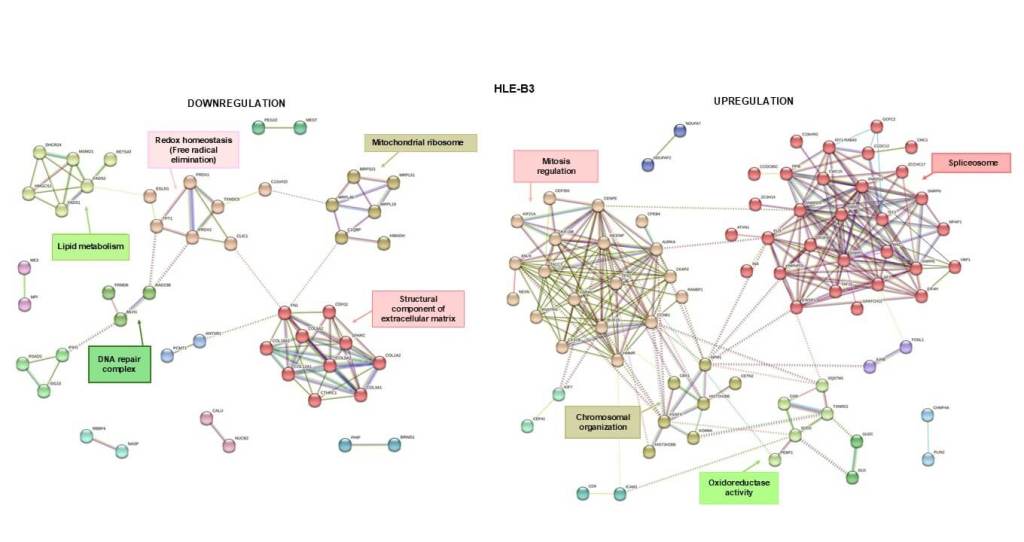
La proteómica no mira una única pista de la escena del crimen: mira toda la escena. Estudia redes enteras de proteínas para entender cómo una enfermedad afecta al organismo, revelando así patrones de proteínas relacionados no solo con los síntomas, sino también con los mecanismos ocultos detrás del desarrollo de la enfermedad (Figura 3).
Retos de la proteómica
Aunque la proteómica ha avanzado significativamente en los últimos años, persisten numerosos desafíos. Entre ellos está la validación clínica de los biomarcadores identificados, utilizando grandes grupos de muestras biológicas, y la estandarización de métodos entre los distintos laboratorios. Sin embargo, su potencial para lo que se conoce como medicina personalizada —enfoque que adapta los tratamientos a las características individuales de cada paciente o grupo de pacientes— es inmenso, especialmente en enfermedades complejas como la enfermedad de Alzheimer o distintos tipos de cáncer.
La proteómica, en definitiva, no solo aporta herramientas diagnósticas innovadoras, sino que también abre puertas a nuevos tratamientos basados en la modulación de proteínas clave en enfermedades crónicas.
Referencias
- Montero-Calle A, Garranzo-Asensio M, Poves C, Sanz R, Dziakova J, Peláez-García A, de los Ríos V, Martínez-Useros J, Fernández-Aceñero MJ, Barderas R. In-Depth Proteomic Analysis of Paraffin-Embedded Tissue Samples from Colorectal Cancer Patients Revealed TXNDC17 and SLC8A1 as Key Proteins Associated with the Disease. Journal of Proteome Research. (2024). doi: 10.1021/acs.jproteome.3c00749.
- Montero-Calle A, Coronel R, Garranzo-Asensio M, Solís-Fernández G, Rábano A, de Los Ríos V, Fernández-Aceñero MJ, Mendes ML, Martínez-Useros J, Megías D, Moreno-Casbas MT, Peláez-García A, Liste I, Barderas R. Proteomics analysis of prefrontal cortex of Alzheimer’s disease patients revealed dysregulated proteins in the disease and novel proteins associated with amyloid-β pathology. Cell Mol Life Sci. (2023). doi: 10.1007/s00018-023-04791-y.
- Rejas-González R, Montero-Calle A, Pastora Salvador N, Crespo Carballés MJ, Ausín-González E, Sánchez-Naves J, Pardo Calderón S, Barderas R, Guzman-Aranguez A. Unraveling the nexus of oxidative stress, ocular diseases, and small extracellular vesicles to identify novel glaucoma biomarkers through in-depth proteomics. Redox Biology. (2024). doi: 10.1016/j.redox.2024.103368
- Rejas-González R, Montero-Calle A, Valverde A, Salvador NP, Carballés MJC, Ausín-González E, Sánchez-Naves J, Campuzano S, Barderas R, Guzman-Aranguez A. Proteomics Analyses of Small Extracellular Vesicles of Aqueous Humor: Identification and Validation of GAS6 and SPP1 as Glaucoma Markers. International Journal of Molecular Sciences, 2024. doi: 10.3390/ijms25136995

Ana Montero Calle
Doctorado en Bioquímica, Biología Molecular y Biomedicina


Rodrigo Barderas Manchado
Doctor en Ciencias Químicas

Cortesía de Muy Interesante
Dejanos un comentario:
