El ARN mensajero (ARNm) es una de las moléculas más importantes de la biología: actúa como intermediario entre el ADN y las proteínas, transmitiendo las instrucciones que permiten a las células fabricar las piezas que las mantienen vivas. No solo se trata de una molécula clave para la vida, sino que también está en el centro de algunos de los desarrollos médicos más recientes, como las vacunas de ARNm contra la COVID-19. Su estructura tridimensional, formada por una serie de pliegues y emparejamientos entre nucleótidos, influye directamente en su estabilidad y en la eficacia con la que es traducido en proteínas. Sin embargo, predecir esa estructura es extremadamente complicado cuando la molécula es larga.
Hasta ahora, los modelos clásicos e incluso la inteligencia artificial podían analizar secuencias largas, pero solo con atajos: por ejemplo, eliminando de los cálculos ciertas formas complejas llamadas pseudonudos. Estos son bucles y cruces en la molécula que implican interacciones más intrincadas que los pliegues comunes, pero que son esenciales para entender su comportamiento real. El nuevo trabajo de IBM, Moderna y Fermioniq presenta un cambio importante: por primera vez, un ordenador cuántico ha simulado la estructura secundaria de un ARNm de 60 nucleótidos incluyendo estos elementos complejos, algo que antes quedaba fuera del alcance de la IA.
Un reto que crece de forma explosiva
En el estudio, los autores señalan que “predecir estructuras con pseudonudos se clasifica como un problema NP-completo”, es decir, un tipo de reto matemático que se vuelve prácticamente intratable conforme aumenta el número de elementos a considerar. La razón es que el número de posibles configuraciones crece de manera exponencial con cada nucleótido añadido. Esto significa que un pequeño incremento en la longitud de la molécula dispara la cantidad de cálculos necesarios, hasta un punto en que los ordenadores convencionales no pueden manejarlo sin recurrir a simplificaciones drásticas.
La investigación utiliza un enfoque cuántico para sortear esa barrera. A diferencia de un bit clásico, que solo puede valer 0 o 1, un cúbit puede estar en una superposición de ambos estados, lo que permite explorar muchas posibilidades al mismo tiempo. Este paralelismo cuántico es especialmente valioso para problemas como la predicción de pliegues moleculares, en los que hay que evaluar enormes cantidades de configuraciones posibles para identificar las más estables.
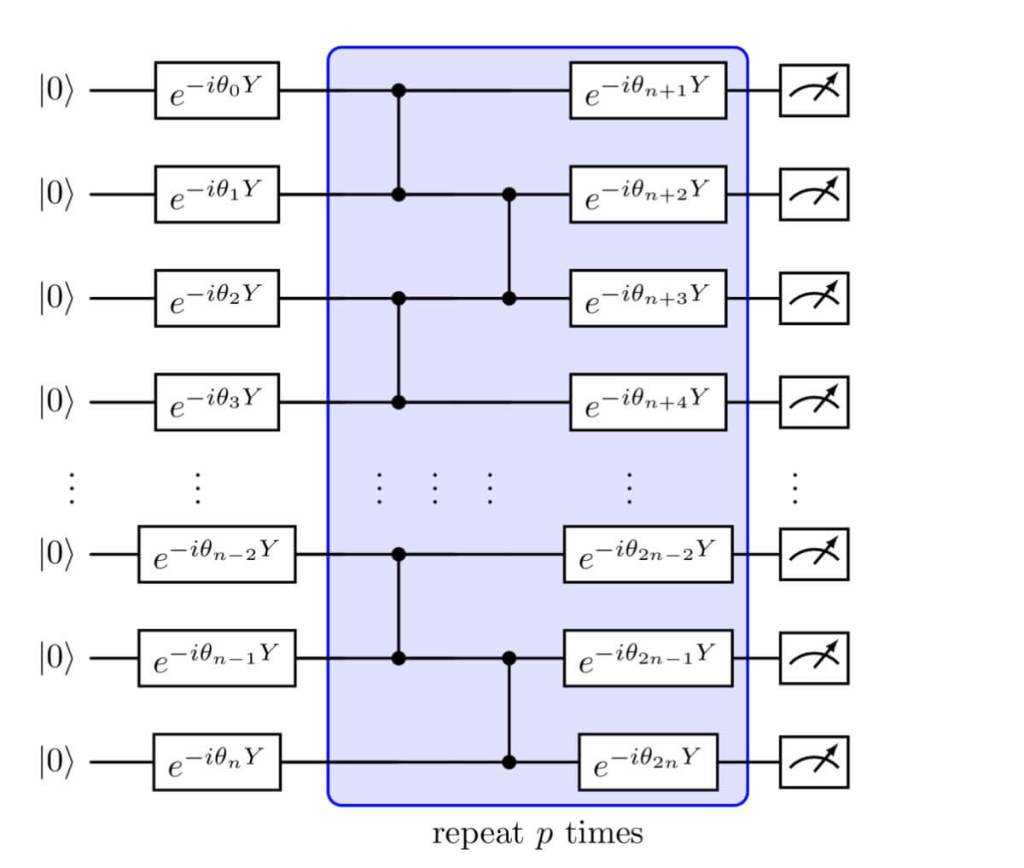
De los 42 a los 60 nucleótidos: un salto histórico
El trabajo se basa en un marco de optimización llamado QUBO (Quadratic Unconstrained Binary Optimization), que traduce el problema de encontrar la estructura más estable en una forma matemática que el ordenador cuántico puede manejar. En un estudio anterior, el equipo ya había logrado simular cadenas de hasta 42 nucleótidos, lo que era entonces el récord. Esta vez, gracias a un nuevo algoritmo y a mejoras en el hardware, llegaron a 60 nucleótidos y 156 cúbits, utilizando procesadores cuánticos de IBM como Heron r2.
Para conseguirlo, emplearon dos estrategias. La primera fue un algoritmo variacional cuántico basado en Conditional Value at Risk (CVaR), una técnica adaptada de la evaluación de riesgos financieros que, aplicada aquí, permite centrarse en las configuraciones de menor energía. La segunda fue un esquema basado en circuitos cuánticos polinomiales instantáneos (IQP), entrenados de forma clásica pero ejecutados en hardware cuántico para la parte de muestreo. En palabras de los autores, este planteamiento permite “delegar en el ordenador cuántico las tareas que son clásicamente difíciles”.

Por qué los pseudonudos son un obstáculo tan duro
Un pseudonudo se produce cuando partes de la molécula que no están adyacentes se aparean de forma que sus bucles se entrecruzan. Esto genera interacciones de largo alcance y una arquitectura espacial más cerrada. Los métodos tradicionales de predicción, como los algoritmos de Zuker o herramientas como ViennaRNA, suelen evitarlos porque su cálculo exacto requiere analizar demasiadas combinaciones. El problema se agrava por el hecho de que el ARNm tiene seis ángulos de torsión en su esqueleto —frente a solo dos en las proteínas—, lo que multiplica aún más la complejidad.
Al incluir los pseudonudos en la simulación, los investigadores se acercan mucho más a las condiciones reales que tendría la molécula en un entorno biológico. Esto no solo mejora la exactitud de las predicciones, sino que también abre la puerta a diseñar ARNm con propiedades específicas para vacunas o terapias. Por ejemplo, una estructura secundaria más estable podría prolongar la vida útil de la molécula dentro del cuerpo, aumentando su eficacia.
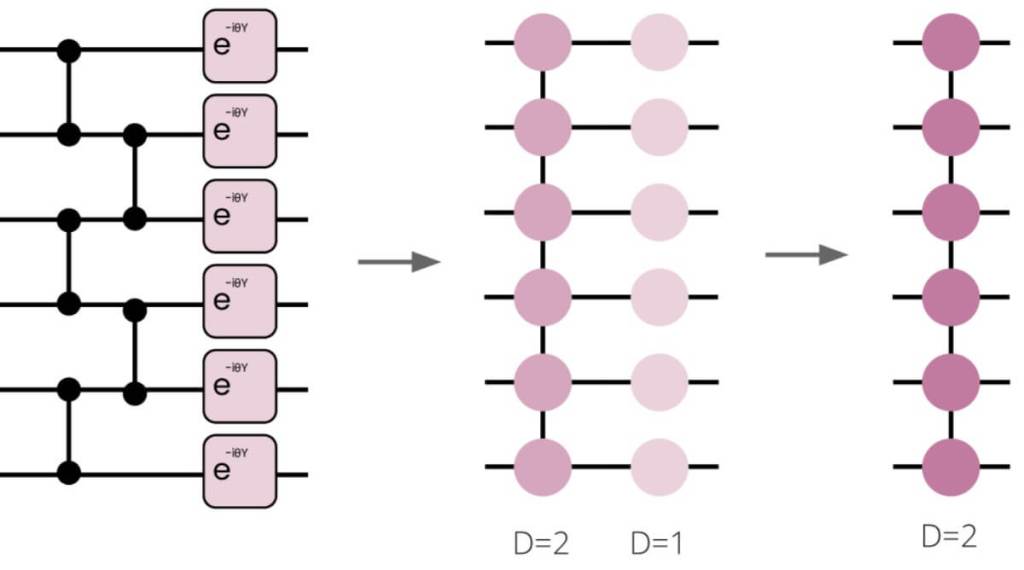
Innovaciones para combatir el ruido cuántico
Uno de los grandes retos de la computación cuántica actual es el ruido, es decir, los errores introducidos por imperfecciones en las operaciones y en las mediciones. El estudio incorpora varias técnicas para mitigar este problema. Entre ellas, destaca la transformación de gauge, que consiste en reconfigurar el problema de manera que los errores típicos del hardware afecten menos al resultado. También aplicaron un umbral de parámetros, que reduce a cero aquellos valores demasiado pequeños para tener un efecto real en la simulación, y una fase de búsqueda local clásica para refinar las soluciones obtenidas por el hardware cuántico.
El resultado de combinar estos pasos fue que, incluso con hardware ruidoso, las simulaciones se acercaron notablemente a la solución óptima. En algunos casos, el postprocesado final consiguió exactamente la estructura más estable prevista por cálculos teóricos, confirmando la utilidad del enfoque híbrido.
Simulaciones más allá del hardware actual
Además de las pruebas en ordenadores cuánticos reales, el equipo utilizó simulaciones de redes tensoriales para explorar cómo se comportaría el algoritmo en máquinas más grandes y sin ruido. Gracias a estas técnicas, pudieron validar la escalabilidad de su enfoque hasta 354 cúbits, lo que equivale a moléculas aún más largas. Aunque este resultado se obtuvo en entornos ideales, sirve como hoja de ruta para cuando el hardware alcance ese nivel de capacidad.
Los experimentos también muestran que no basta con aumentar el número de capas en los circuitos cuánticos: hay que ajustar cuidadosamente la forma en que se entrenan para evitar que el optimizador se pierda en espacios de parámetros demasiado amplios. Estrategias como la optimización por capas incrementales demostraron mejorar la convergencia sin aumentar el coste computacional de forma desproporcionada.
Implicaciones para la biomedicina
La posibilidad de predecir de forma más exacta la estructura secundaria del ARNm podría acelerar el diseño de vacunas y terapias génicas. Las vacunas de ARNm actuales, como las de Pfizer-BioNTech y Moderna contra la COVID-19, dependen de que la molécula llegue intacta y activa a las células. Un conocimiento más preciso de cómo se pliega y qué factores afectan a su estabilidad permitiría optimizar las secuencias para que resistan mejor las condiciones del organismo o para que se traduzcan más eficientemente en la proteína deseada.
Más allá de las aplicaciones médicas, este trabajo es un ejemplo de cómo la computación cuántica empieza a resolver problemas científicos reales que no son accesibles a la IA convencional. Aunque todavía falta para que esta tecnología sea habitual en laboratorios y empresas, cada nuevo avance práctico aumenta su relevancia y demuestra que no se trata solo de un experimento académico.
Un paso, no el final del camino
Los propios autores advierten que, para ir más allá, será necesario desarrollar técnicas avanzadas para integrar estos circuitos específicos en el hardware cuántico existente, reduciendo la sobrecarga de compuertas y mejorando la tolerancia a errores. También planean explorar métodos no variacionales, como algoritmos inspirados en campos magnéticos sesgados o cadenas de Markov mejoradas con cuántica, que podrían ofrecer ventajas en ciertos tipos de problemas.
Este avance marca un hito porque combina un desafío biológico complejo con un enfoque computacional emergente, logrando un resultado tangible que supera las limitaciones previas. El salto de 42 a 60 nucleótidos en un ordenador cuántico no solo es una cifra: es una demostración de que la colaboración entre la biotecnología y la física cuántica puede abrir nuevas fronteras en la investigación aplicada.
Referencias
- Vaibhaw Kumar, Dimitris Alevras, Mihir Metkar, Eline Welling, Chris Cade, Ido Niesen, Triet Friedhoff, Jae-Eun Park, Saurabh Shivpuje, Mariana LaDue, Wade Davis, Alexey Galda. Towards secondary structure prediction of longer mRNA sequences using a quantum-centric optimization scheme. arXiv, 2025. doi:10.48550/arXiv.2505.05782.
Cortesía de Muy Interesante
Dejanos un comentario:

